陈棣成1,#, 胡琬祺1,#, 刘慧婷1, 周毅荣1,邱天予1,黄奕晖1,王孜1,林美金2, 林良杰3, 武志刚3, 王家正3, 陈浩4, 陈希5, 延根6,郭迪7,林建忠8,屈小波1,*
1厦门大学, 电子科学系, 电子科学与技术学院, 中国, 厦门;
2厦门大学, 海洋与地球科学学院应用海洋物理与工程系, 中国, 厦门;
3飞利浦医疗, 中国, 北京;
4上海交通大学, 生物医学工程学院, 中国, 上海;
5哈佛医学院, 麦克莱恩医院, 美国, 贝尔蒙特;
6厦门医学院附属第二医院, 放射科, 厦门;
7厦门理工学院, 计算机与信息工程学院, 中国, 厦门;
8厦门大学附属中山医院, 放射科, 中国, 厦门;
*通信作者邮箱: quxiaobo <at> xmu.edu.cn
引用
D. Chen, W. Hu, H. Liu, Y. Zhou, T. Qiu, Y. Huang, Z. Wang, M. Lin, L. Lin, Z. Wu, J. Wang, H. Chen, X. Chen, G. Yan, D. Guo, J. Lin, and X. Qu, Magnetic resonance spectroscopy deep learning denoising using few in vivo data, IEEE Transactions on Computational Imaging, DOI: 10.1109/TCI.2023.3267623, 2023.
概要
磁共振波谱(MRS)是一种揭示代谢信息的非侵入性工具。1H-MRS技术的一个挑战是低信噪比(SNR)[1-2]。为了提高信噪比,一个典型的方法是大量的重复采集,然后对其求均,简称信号平均(SA)[3-4]。而数据的重复采集时间与SA成正比。在SA为128的常规临床扫描下,完整的MRS大约需要10分钟。另外,最近有学者引入深度学习来提高信噪比,但这些深度学习方法一般只使用模拟数据来训练。这可能会阻碍MRS的应用,因为模拟数据没有考虑实际中一些不理想的条件,如采集系统的缺陷,以及被试的生理活动或心理状态等。
主要内容
有别于物理智能产生大量合成数据[5],本文提出了一种纯利用部分实际数据重复采样的新方案。设计了一种深度学习模型—Refusion Long-Short-Term Memory (ReLSTM),学习从低信噪比(24 SA)到高信噪比(128 SA)的MRS时域数据的去噪映射。为了训练该深度模型,构建了一个由少量25个健康人脑额叶波谱组成训练集。其中,每个波谱数据通过从128次重复采集中随机选择24个样本平均作为低信噪比输入,随机选择124个样本平均作为高信噪比标签,来获得大量的训练真实训练数据。因此,我们把少量的25个波谱巧妙地扩增成25000个(25×1000)波谱数据进行网络训练。
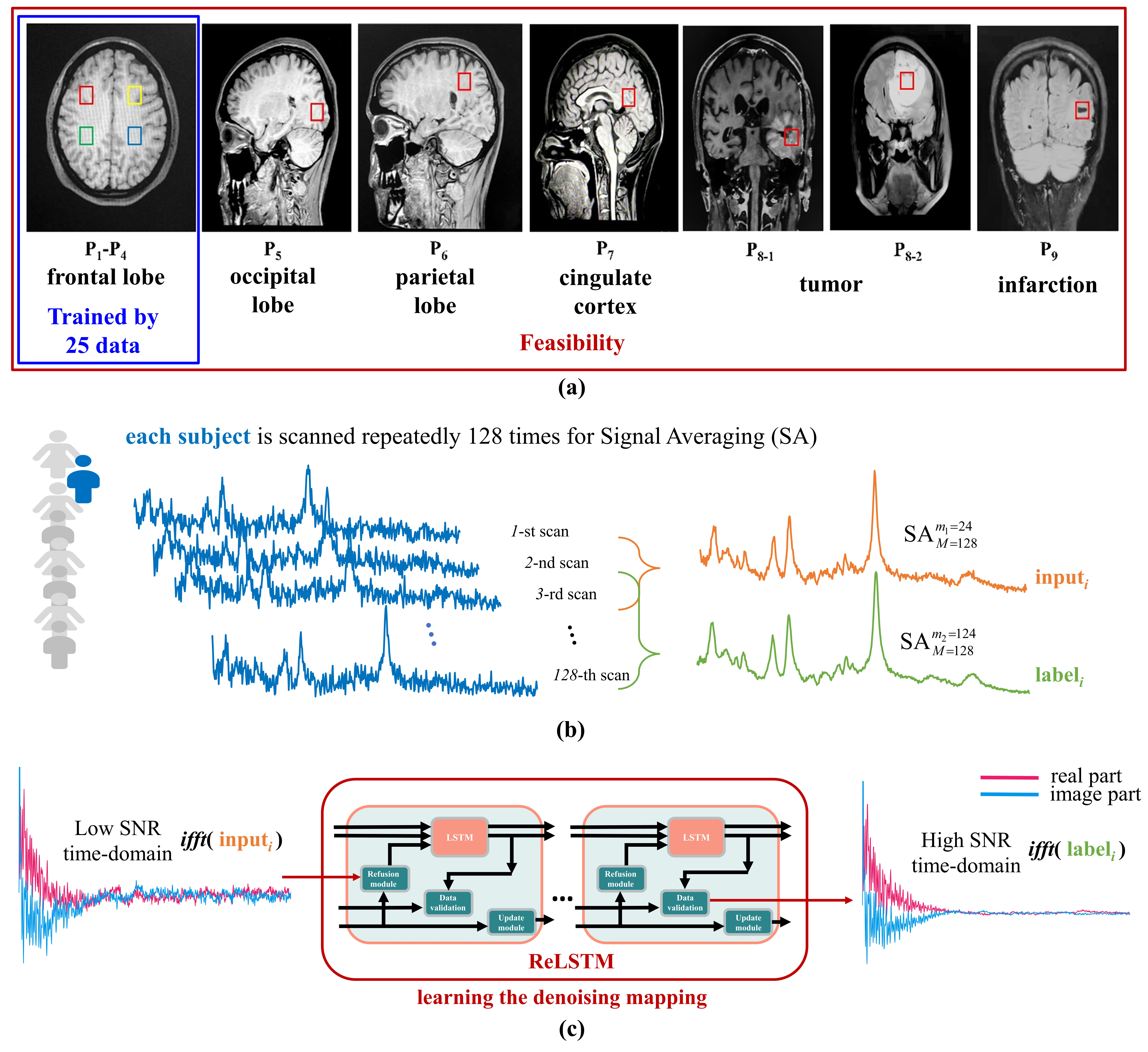
另外,在可行性检验中,我们不仅选取了额叶(P1-4)的波谱,还选取了不同受试者的其他位置的波谱,如枕叶、顶叶、扣带皮层(P5-P7),甚至病变区(P8-P9),共1000份数据。所有的实施步骤见图1所示。

图 1. ReLSTM深度学习波谱去噪的流程。(a)数据概述:25个健康额叶波谱用于训练ReLSTM, 不同人的4个额叶波谱,3个其他健康脑区波谱,3个病变区域波谱用于可行性检验。(b)生成一个训练对实例:从128次重复采集中随机选择24个样本平均作为低信噪比输入,随机选择124个样本平均作为高信噪比标签。 (c)深度去噪网络ReLSTM概述:学习从低信噪比(24 SA)到高信噪比(128 SA)的MRS时域数据的去噪映射。其中ifft表示离散傅里叶反变换,把波谱转换到对应的时域信号进行处理。
可行性检验表明,仅为常规设置的20%的情况下(SA为24),用ReLSTM去噪后的谱可以得到代谢物浓度的估计,其可靠性与128 SA获得的高信噪比谱相当,如表1所示。此外,与先进的Low-Rank[6]去噪方法相比,ReLSTM在量化一些重要生物标志物方面实现了更低的相对误差和Cramér-Rao下界。更多细节查阅全文:
全文链接:https://dx.doi.org/10.1109/TCI.2023.3267623
预印本链接:https://arxiv.org/abs/2101.11442
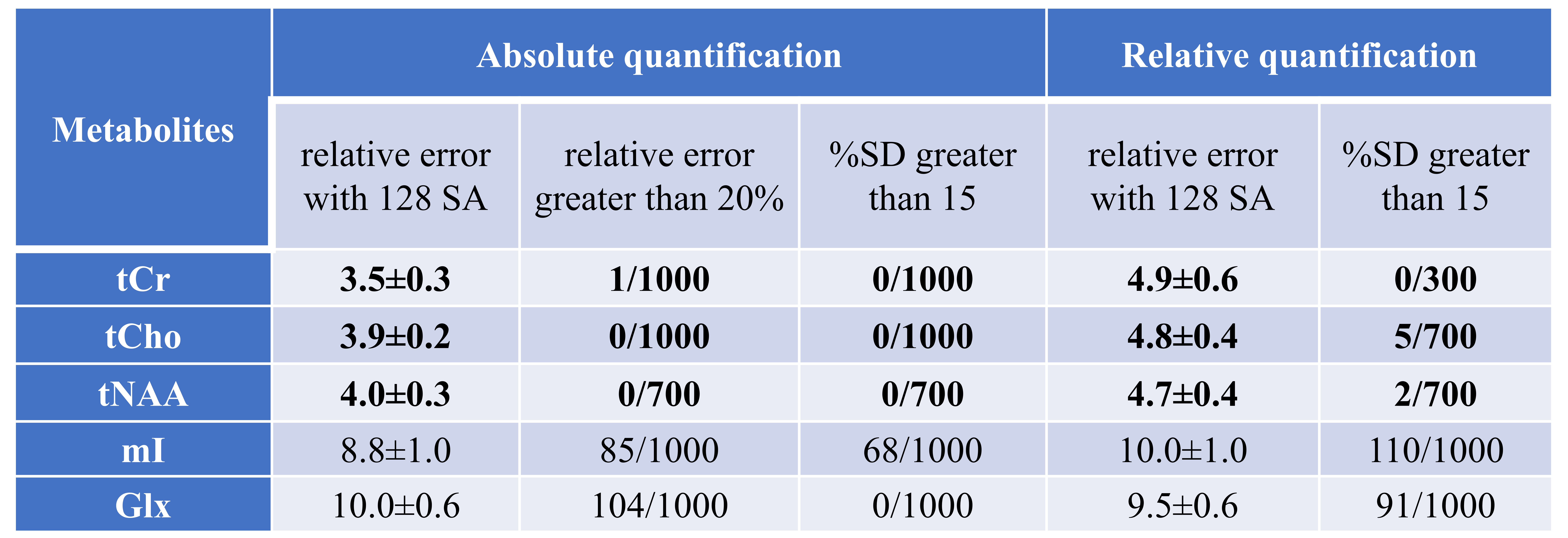
表1. ReLSTM去噪结果的代谢物浓度评估(与128 SA(高信噪比)波谱比较)。
资源
CloudBrian-MRS link: https://csrc.xmu.edu.cn/CloudBrain.html
致谢
该工作受到了国家优青等重要项目支持,得到了国家自然科学基金(62122064, 61971361, 61871341, 61811530021, 81401405)、福建省自然科学基金(2021J011184)、福建省健康教育联合研究项目(2019-WJ-31)和厦门大学南强拔尖人才计划资助。
参考文献
[1] J.B. Poullet, D.M. Sima, and S. Van Huffel, "MRS signal quantitation: A review of time-and frequency-domain methods," Journal of Magnetic Resonance, vol. 195, no. 2, pp. 134-144, 2008.
[2] S.W. Provencher, "Estimation of metabolite concentrations from localized in vivo proton NMR spectra," Magnetic Resonance in Medicine, vol. 30, no. 6, pp. 672-679, 1993.
[3] J. Near, A.D. Harris, C. Juchem, R. Kreis, M. Marjańska, G. Öz, J. Slotboom, M. Wilson, and C. Gasparovic, "Preprocessing, analysis and quantification in single-voxel magnetic resonance spectroscopy: Experts' consensus recommendations," NMR in Biomedicine, vol. 34, no. 5, pp. 4257, 2020.
[4] C. Choi et al., "Measurement of glycine in the human brain in vivo by 1H‐MRS at 3 T: Application in brain tumors," Magnetic Resonance in Medicine, vol. 66, no. 3, pp. 609-618, 2011.
[5] Q. Yang, Z. Wang, K. Guo, C. Cai and X. Qu, "Physics-Driven Synthetic Data Learning for Biomedical Magnetic Resonance: The imaging physics-based data synthesis paradigm for artificial intelligence," IEEE Signal Processing Magazine, vol. 40, no. 2, pp. 129-140, 2023.
[6] T. Qiu et al., "An automatic denoising method for NMR spectroscopy based on low-rank Hankel model," IEEE Transactions on Instrumentation & Measurement, vol. 70, pp. 1-12, 2021.