Low Rank Enhanced Matrix Recovery of Hybrid Time and Frequency Data in Fast Magnetic Resonance Spectroscopy (中文,English)
鲁恒发1, 张心林1, 邱天予1, 杨健1, 应佳熙1, 郭迪2, 陈忠1, 屈小波1,#
1 厦门大学,电子科学系,福建等离子体与磁共振重点研究实验室,中国,厦门;
2 厦门理工学院,计算机与信息工程学院,中国,厦门。
# Emails: quxiaobo <at> xmu.edu.cn or quxiaobo2009 <at> gmail.com
引用: Hengfa Lu, Xinlin Zhang, Tianyu Qiu, Jian Yang, Jiaxi Ying, Di Guo, Zhong Chen, Xiaobo Qu, Low rank enhanced matrix recovery of hybrid time and frequency data in fast magnetic resonance spectroscopy, IEEE Transactions on Biomedical Engineering, 65(4): 809-820, 2018.
摘要:
磁共振波谱(Magnetic Resonance Spectroscopy,MRS)是生物医学工程中常用 的分析物质化学成分与生物蛋白质结构的手段之一。多维MRS 减少了谱峰的拥挤和重叠且提供了新的原子核之间相互关系的信息,对复杂生物大分子分析有重要意义,但受限于间接维演化等物理机制,典型多维谱的数据采集时间随维度和分辨率的增加而急剧增加。因此,降低多维MRS的采样时间非常重要。
时空编码是通过物理实验序列实现快速MRS 的典型方法之一,该技术通过并行编码间接维的频率信息实现信号快速采集,但该技术较强的数据采集梯度容易对仪器造成伤害。为降低对梯度强度要求,非均匀采样被引入到时空编码MRS中,但同时也使得混合时间-频率平面的信号缺失。 从信号处理来看,如何对缺失的混合时间-频率信号进行高质量重建是一个挑战性问题。当前最前沿方法主要是通过约束频谱的稀疏性来重建时空编码数据,但这种方法可能导致谱峰失真甚至丢失,因此亟待开发新的信号重建方法。
本文主要从MRS 时间信号的低秩特性出发,研究如何将混合时间-频率信号建模成二维指数函数并进行缺失数据重建。提出一种基于低秩分块Hankel 矩阵的混合时间-频率平面信号重建模型,并推导了其快速数值算法。实验结果表明,所提方法重建的谱峰线型比频谱稀疏方法的重建谱峰更接近真实谱峰,可以更准确地量化物质中不同基团的质子共振体积,量化准确性提升达5 倍以上。本文所提方法可以拓展到MRS 的去噪等其它重建问题,甚至拓展到任意二维 指数的重建。
方法:
1. 背景
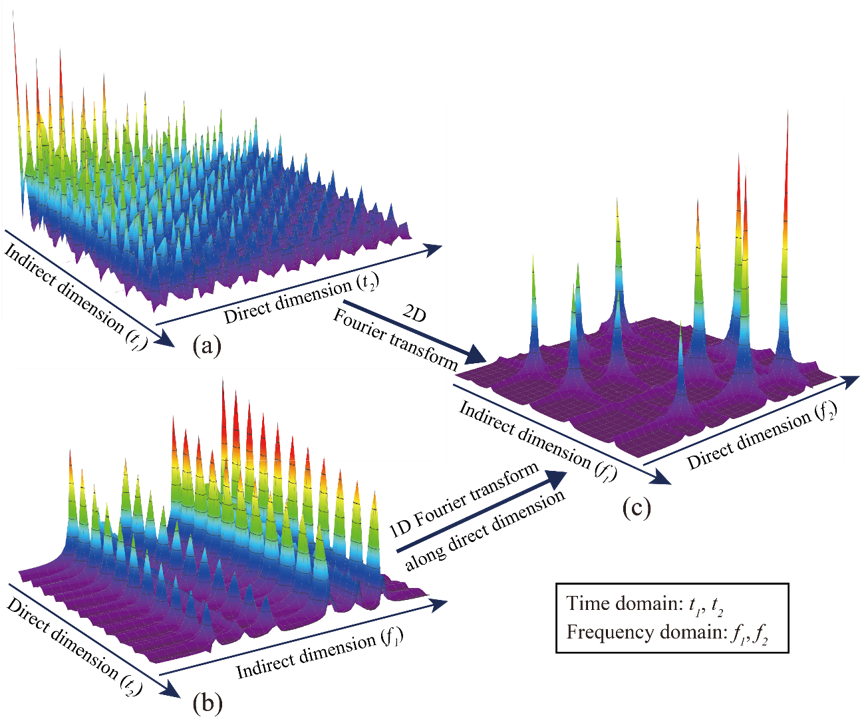
在时空编码波谱中非均匀采样技术可以减缓实验对高强度梯度的需求。与传统波谱实验不同,时空编码波谱实验特定的编码方式会使得采集的信号其直接维位于时间域,间接维位于频率域,即信号位于混合时间-频率域 (图1(b))。因此,只需对采集到的时空编码信号进行一维傅里叶变换即可得到完整的二维波谱。但,非均匀采样技术的引入使得数据点在混合时间-频率域丢失(图2)。因此,为了获得高分辨的波谱需要开发合适的波谱重建方法。

图 1 传统二维波谱与时空编码二维波谱示意图。(a) 传统波谱实验获取的时间-时间域信号;(b) 时空编码实验获取的混合时间-频率域信号;(c) 二维频率-频率域波谱信号。
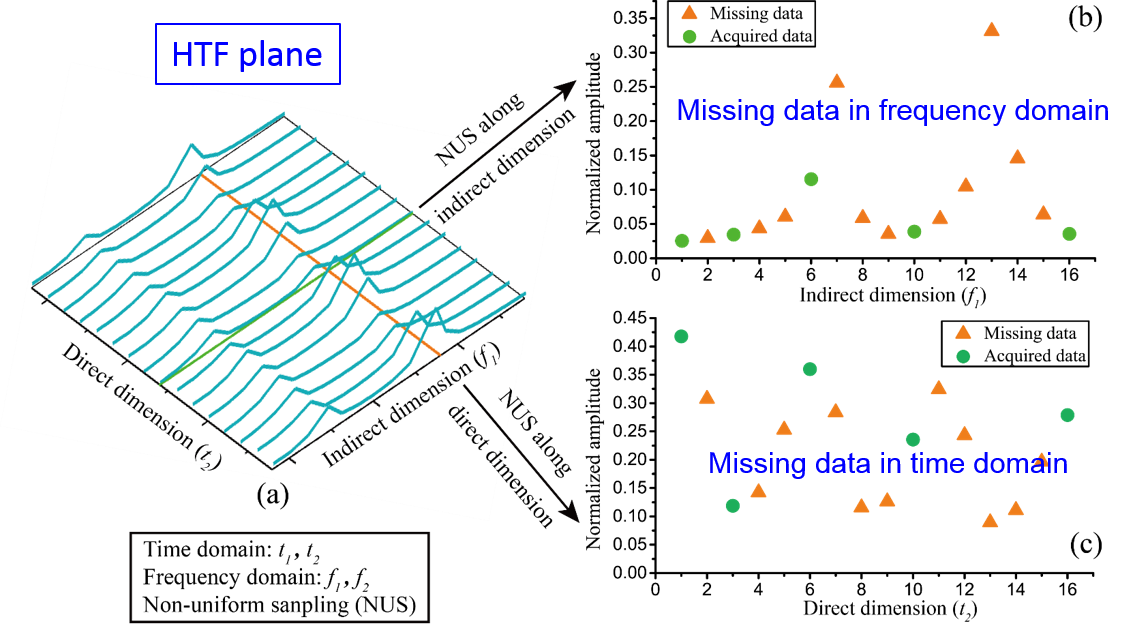
图 2 混合时间-频率平面示意图。
2. 低秩分块Hankel矩阵重建方法
二维 MRS 信号可以建模为一系列衰减型指数信号的叠加 [12]。研究表明,由有限个二维指数信号构建的分块Hankel矩阵是低秩矩阵。基于此性质,我们提出了基于MRS信号低秩的混合时间-频率信号恢复模型:
$$ \min_\mathbf{G} \left \| \mathbf{BF}_\textit{freq}^{-1}\mathbf{G} \right \|_*+\frac{\lambda }{2}\left \| \mathbf{Y-P}_\Omega \mathbf{G} \right \|_F^2, $$
其中,$\mathbf{G}$ 表示待重建的二维混合时间-频率域信号;$\mathbf{Y}$表示对采集信号进行非采样点位置填零的信号;$ \mathbf{P}_\Omega$表示混合时间-频率平面的非均匀采样算子,该算子会对非采样的数据点位置进行填零操作;$\left \| \cdot \right \|_F$表示矩阵的F范数(元素平方和开根号)。参数$\lambda$用于平衡核范数项$\left \| \mathbf{BF}_\textit{freq}^{-1}\mathbf{G} \right \|_*$和数据校验项$\left \| \mathbf{Y-P}_\Omega \mathbf{G} \right \|_F^2$。本质上,该模型希望从采集到的数据中找到谱峰个数最少的波谱。
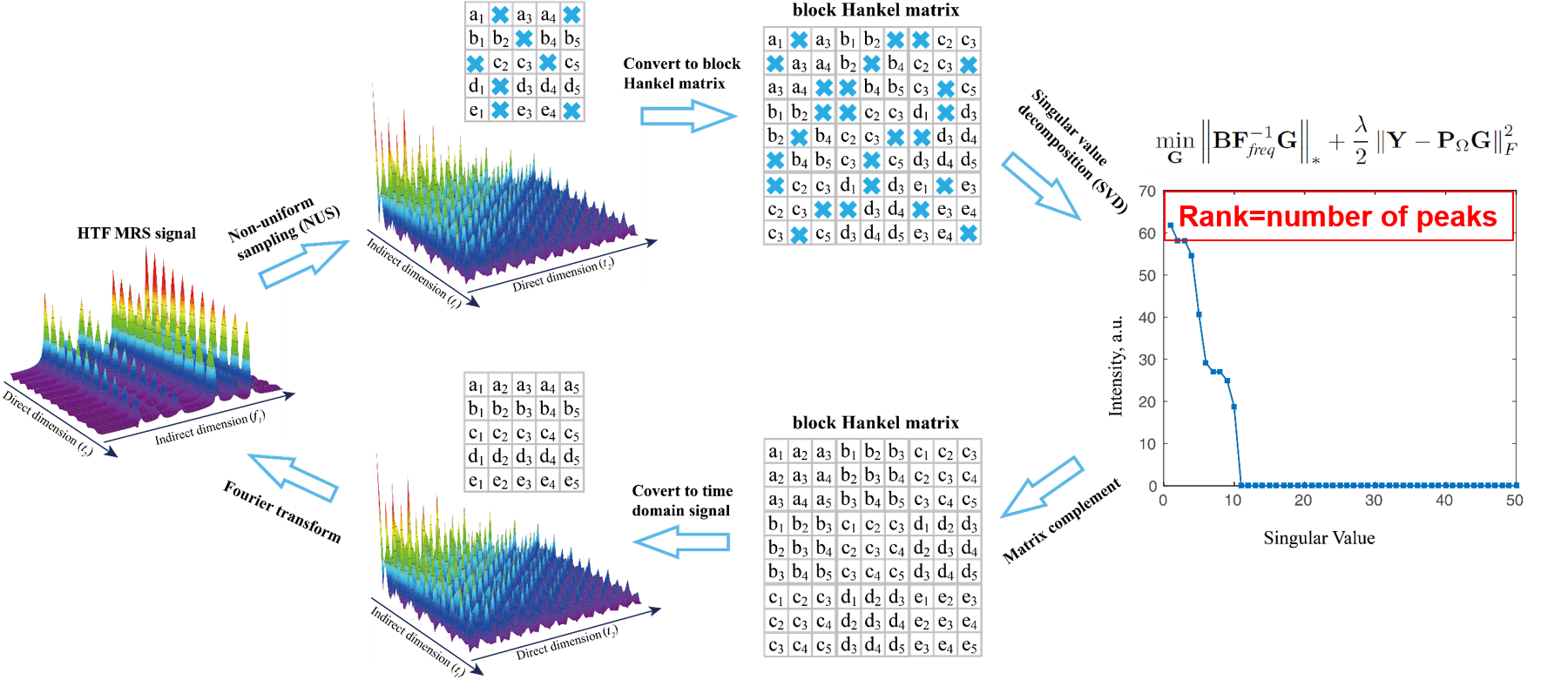
图 3 低秩分块Hankel矩阵波谱重建方法流程图
3. 主要结果
我们使用油脂样品采集全采样的二维时空编码相关谱(Correlation Spectroscopy, COSY)对所提方法进行验证。这种油脂存在肝脏脂肪中,使用MRS对脂肪含量的测量在临床应用中具有重要的应用价值。图4展示了重建的时空编码波谱。基于波谱稀疏重建的波谱中出现了8个低强度谱峰的强度削弱情况(图4 (b))。相反,所提方法提供了这8个谱峰的可靠重建(图4 (c))。在随所有谱峰进行重建谱峰和全采样谱峰强度相关性统计中,基于波谱稀疏方法和所提方法谱峰强度相关性系数R2均接近1,这意味着两种方法均提供了很好的重建结果。但,在只统计低强度谱峰相关性情况下(归一化谱峰强度从0到0.25),基于波谱稀疏方法的相关性系数下降到0.9966 (图5 (a)和(c)),但所提方法谱峰相关系数仍有0.9998 (图5 (b)和(d)). 谱峰相关性结果表明所提发方法重建谱峰更接近参考波谱谱峰。
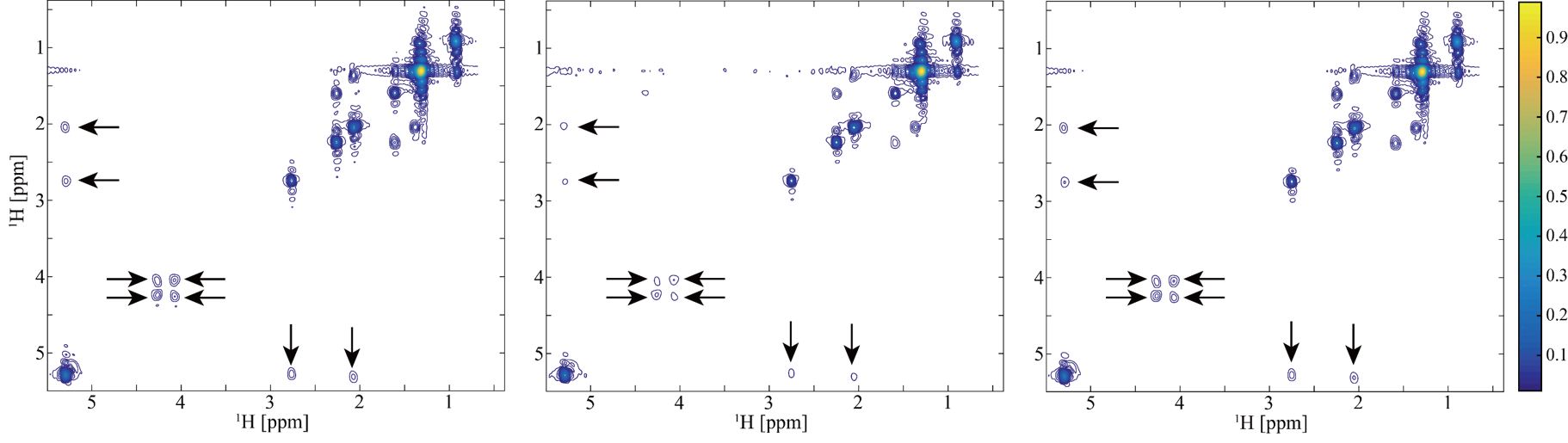
图 4 时空编码COSY波谱重建结果。(a) 全采样波谱;(b) 基于波谱稀疏方法重建波谱;(c) 所提方法重建波谱。注:所有波谱显示范围均为90 (使用MATLAB contour函数)。
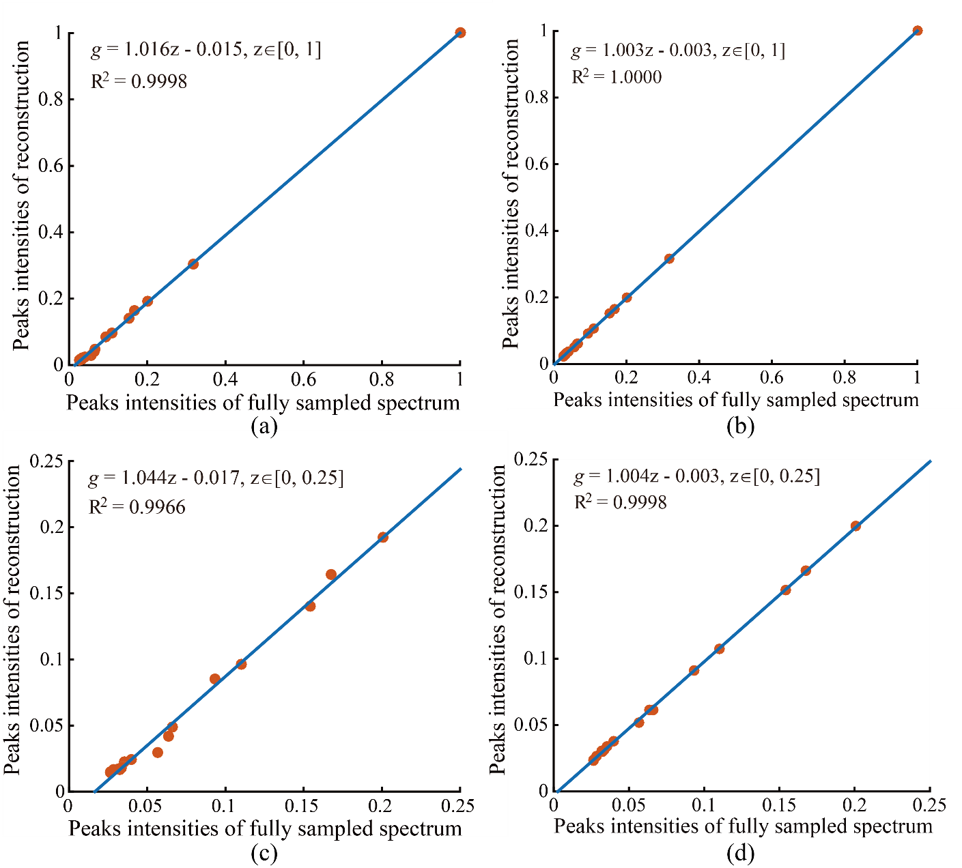
图 5 全采样时空编码波谱与重建波谱谱峰强度相关性分析。(a)和(b)分 别是统计所有谱峰情况下基于波谱稀疏方法和所提方法 的谱峰强度相关性结果;(c)和(d)分 别是在统计谱峰强度从 0 至 0.25 情况下 基于波谱稀疏方法和所提方法 的谱峰强度相关性结果。公式 g = az + b表示使用多项式拟合的全采样波谱 谱峰与重建波谱谱峰强度曲线(蓝色线)。 R2 代表拟合曲线的皮尔逊线性相关系 数。R2 越接近 1,代表全采样波谱与重建波谱的相关性越强。注:图中的波谱信号强度都已经通过除以信号强度的最大绝对值的方式归一化到 [0,1]。只有每个谱峰的最大幅值用于相关性统计分析。
代码下载:
所提方法的MATLAB代码可以从 这里 下载。
致谢:
该工作受到国家自然科学基金 (61571380, 61672335, 61601276, and U1632274),国家重点研发计划 (2017YFC0108703),福建省自然科学基金 (2015J01346 and 2016J05205),厦门市重大疾病研发计划 (3502Z20149032)和中央高校项目(20720150109)的资助。作者感谢林雁勤副教授在时空编码序列方面的宝贵意见,感谢Lucio Frydman教授分享基于波谱稀疏的时空编码数据恢复方法代码,感谢施天谟教授帮忙润色论文,以及审稿人和编辑有建设性的评论。
参考文献:
[1] Y. Shrot and L. Frydman, “Compressed sensing and the reconstruction of ultrafast 2D NMR data: Principles and biomolecular applications,” J. Magn. Resonance, vol. 209, no. 2, pp. 352–358, 2011.
[2] X. Qu et al., “Accelerated NMR spectroscopy with low-rank reconstruction,” Angewandte Chemie Int. Edition, vol. 54, no. 3, pp. 852–854, 2015.
[3] L. Frydman et al., “The acquisition of multidimensional NMR spectra within a single scan,” Proc. Nat. Academy Sci., vol. 99, no. 25, pp. 15858– 15862, 2002.
[4] Y. Hua and T. K. Sarkar, “Matrix pencil method for estimating parameters of exponentially damped/undamped sinusoids in noise,” IEEE Trans. Acoust., Speech, Signal Process., vol. 38, no. 5, pp. 814–824, May 1990.
[5] J.-F. Cai et al., “A singular value thresholding algorithm for matrix completion,” SIAM J. Optim., vol. 20, no. 4, pp. 1956–1982, 2010.
[6] N. Srebro, “Learning with matrix factorizations,” Ph.D. dissertation, Dept. Elect. Eng. Computer Sci., Massachusetts Institute of Technology, Cambridge, MA, USA, 2004.
[7] Y. Hua and T. K. Sarkar, “Matrix pencil method for estimating parameters of exponentially damped/undamped sinusoids in noise,” IEEE Trans. Acoust., Speech, Signal Process., vol. 38, no. 5, pp. 814–824, May 1990.
[8] S. G. Hyberts et al., “Poisson-gap sampling and FM reconstruction for enhancing resolution and sensitivity of protein NMR data,” J. Amer. Chem. Soc., vol. 132, no. 7, pp. 2145–2147, 2010.
[9] J. Ying et al., “Hankel matrix nuclear norm regularized tensor completion for N -dimensional exponential signals,” IEEE Trans. Signal Process., vol. 65, no. 14, pp. 3702–3717, 2017.
[10] H. Lu et al., “A low rank hankel matrix reconstruction method for ultrafast magnetic resonance spectroscopy,” in Proc. 39th Annu. Int. Conf. IEEE Eng. Med. Biol. Soc., 2017, pp. 3267–3272.
[11] Y. Chen and Y. Chi, “Robust spectral compressed sensing via structured matrix completion,” IEEE Trans. Inf. Theory, vol. 60, no. 10, pp. 6576–6601, Oct. 2014.
[12] J. C. Hoch and A. S. Stern, NMR Data Processing. New York, NY, USA: Wiley-Liss, 1996.
[13] K. Kazimierczuk and V. Y. Orekhov, “Accelerated NMR spectroscopy by using compressed sensing,” Angewandte Chemie Int. Edition, vol. 50, no. 24, pp. 5556–5559, 2011.
[14] M. Betz et al., “Biomolecular NMR: A chaperone to drug discovery,” Current Opinion Chem. Biol., vol. 10, no. 3, pp. 219–225, 2006.
[15]
G. Topcu and A. Ulubelen, “Structure elucidation of organic compounds from natural sources using 1D and 2D NMR techniques,” J. Mol. Struct., vol. 834–836, pp. 57–73, 2007.
[16]
A.-H. M. Emwas et al., “NMR-based metabolomics in human disease diagnosis: Applications, limitations, and recommendations,” Metabolomics, vol. 9, no. 5, pp. 1048–1072, 2013.