
基于小样本鲁棒深度学习的快速磁共振成像方法(中文,English)
王孜1, 钱晨1, 郭迪2, 孙鸿伟3, 李如帅4, 赵波5, 屈小波1,*
1 厦门大学,电子科学系,福建等离子体与磁共振重点研究实验室,健康医疗大数据国家研究院,中国,厦门.
2 厦门理工学院,计算机与信息工程学院,中国,厦门.
3 联影智能影像技术研究院,中国,北京.
4 南京市第一医院,核医学系,中国,南京.
5 德克萨斯大学奥斯汀分校,生物医学工程系,美国,奥斯汀.
* Emails: quxiaobo <at> xmu.edu.cn or quxiaobo2009 <at> gmail.com
引用
Zi Wang, Chen Qian, Di Guo, Hongwei Sun, Rushuai Li, Bo Zhao, Xiaobo Qu, One-dimensional Deep Low-rank and Sparse Network for Accelerated MRI, IEEE Transactions on Medical Imaging, DOI: 10.1109/TMI.2022.3203312, 2022.
概要
磁共振成像是一种无创、无辐射的分子影像技术,使解剖结构和生理功能可视化。成像时间长是制约磁共振成像技术发展的根本性问题之一。快速采样能突破奈奎斯特采样准则实现加速成像,但信号缺失会引入图像的严重伪影,因此实现欠采样信号的高保真重建对快速磁共振成像而言至关重要。
近年来,深度学习凭借高质量的信号恢复、超快速的重建速度已经在快速磁共振成像领域展现出惊人的性能。但仍存在许多亟待解决的问题,如内存消耗大、缺乏鲁棒性等,这也极大地限制了其广泛应用。因此,基于磁共振信号采样,设计深度学习算法,实现更低的计算成本和更鲁棒的性能,是本工作的主要出发点。
主要内容
在快速磁共振成像方面,基于磁共振图像或其对应的k空间都是二维的这一事实,目前最先进的深度学习重建方法大多采用二维卷积神经网络。在本工作中,基于磁共振信号采样,我们提出了一种全新的可分离的一维卷积学习方案,使深度神经网络更容易训练和泛化(图1)。进一步地,利用多线圈数据的低秩性和图像变换域的稀疏性,提出了网络参数少、内存高效的一维深度低秩稀疏网络,称为ODLS。在活体膝关节和大脑数据上的大量结果表明,所提方法在视觉和定量方面都比前沿的方法提供了更好的重建性能,同时允许在高度有限的数据上成功地训练深度网络,小样本训练无过拟合情况,克服了大规模数据集匮乏的问题。此外,所提方法对不同的欠采样场景、训练与测试数据之间的不匹配问题也展现了良好的鲁棒性。

图1. 一维可分深度学习流程图。
在活体膝关节和大脑数据的快速成像实验中,相比与其他前沿方法,所提方法ODLS在训练对象人数有限的情况下,拥有出色的伪影抑制和细节保留能力(图2)。这表明了ODLS非常适用于训练对象人数高度有限的情况,能够克服无法获得大规模数据集或需要依赖额外数据扩增的瓶颈问题。
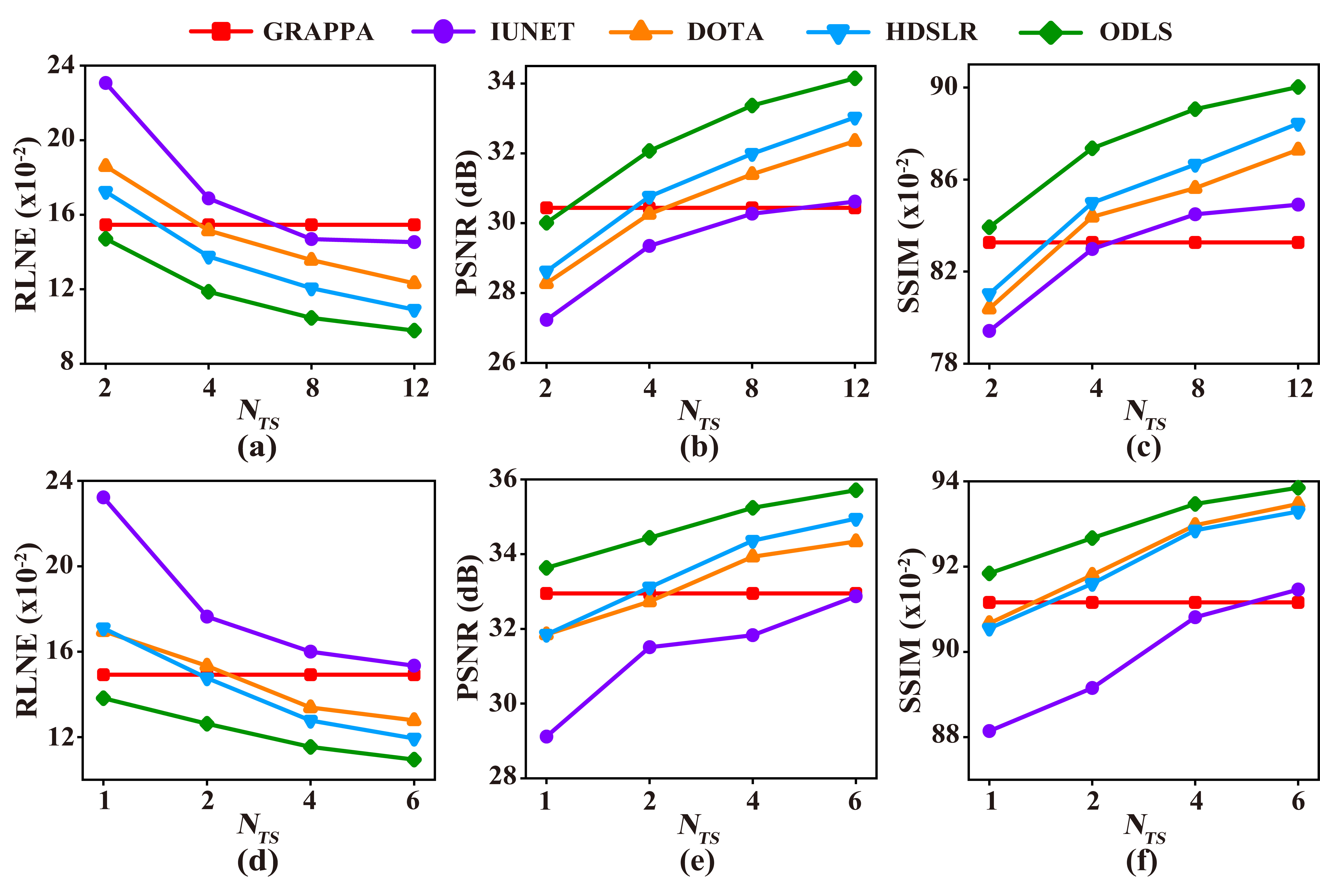
图2. 不同训练对象人数NTS下的膝关节和大脑数据重建结果的定量比较。(a)到(c)分别为膝关节重建的RLNE、PSNR和SSIM。(d)到(f)分别为大脑重建的RLNE、PSNR和SSIM。注:使用4倍加速一维笛卡尔欠采样。
限制现有深度学习方法广泛应用于快速磁共振成像的一个关键因素是缺乏鲁棒性。在实际应用中,它们很难克服训练数据和测试数据之间的不匹配,这意味着需要花费大量的时间对网络进行重新训练来处理不同场景下的各种任务。在这里,我们重点关注膝关节解剖方向、大脑对比度、解剖部位不匹配这三大类问题。实验结果表明,ODLS在不匹配重建下,依旧保持了高质量的重建性能,可以很好地抑制伪影并保留图像细节。而对比的前沿深度学习方法均出现了明显伪影和图像结构丢失(图3)。这表明了ODLS针对常见的不匹配情况具有良好的鲁棒性,可以在广泛的场景下有效工作。
更多ODLS在不同的欠采样场景、不匹配场景和高维可分重建扩展上的优异表现详见全文。
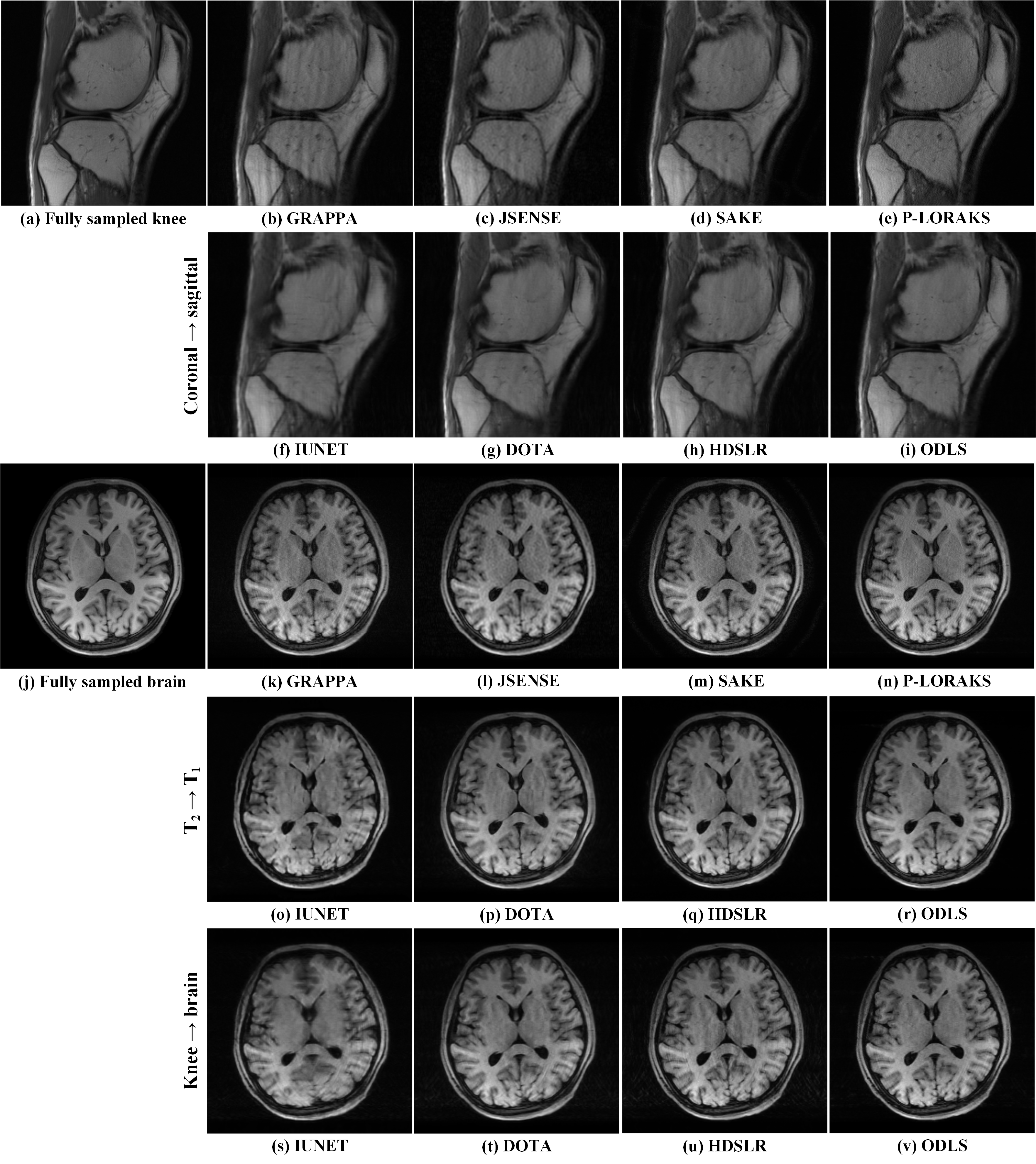
图3. 膝关节解剖方向、大脑对比度、解剖部位不匹配下的重建结果。注:使用4倍加速一维笛卡尔欠采样。
相关资料
致谢
本工作得到了国家自然科学基金(62122064、61971361、61871341),福建省自然科学基金(2021J011184),厦门大学校长基金(0621ZK1035)和厦门大学南强拔尖人才计划的资助。
参考文献
[1] M. Lustig, D. Donoho, and J. M. Pauly, “Sparse MRI: The application of compressed sensing for rapid MR imaging,” Magn. Reson. Med., vol. 58, no. 6, pp. 1182-1195, 2007.
[2] K. P. Pruessmann, M. Weiger, M. B. Scheidegger, and P. Boesiger, “SENSE: Sensitivity encoding for fast MRI,” Magn. Reson. Med., vol. 42, no. 5, pp. 952-962, 1999.
[3] M. A. Griswold et al., “Generalized autocalibrating partially parallel acquisitions (GRAPPA),” Magn. Reson. Med., vol. 47, no. 6, pp. 1202-1210, 2002.
[4] Z. Liang, “Spatiotemporal imaging with partially separable functions,” in 4th IEEE International Symposium on Biomedical Imaging (ISBI), 2007, pp. 988-991.
[5] Y. Yang, F. Liu, Z. Jin, and S. Crozier, “Aliasing artefact suppression in compressed sensing MRI for random phase-encode undersampling,” IEEE Trans. Biomed. Eng., vol. 62, no. 9, pp. 2215-2223, 2015.
[6] X. Zhang et al., “A guaranteed convergence analysis for the projected fast iterative soft-thresholding algorithm in parallel MRI,” Med. Image Anal., vol. 69, 101987, 2021.
[7] X. Zhang et al., “Accelerated MRI reconstruction with separable and enhanced low-rank Hankel regularization,” IEEE Trans. Med. Imaging, DOI: 10.1109/TMI.2022.3164472, 2022.
[8] P. J. Shin et al., “Calibrationless parallel imaging reconstruction based on structured low-rank matrix completion,” Magn. Reson. Med., vol. 72, no. 4, pp. 959-970, 2014.
[9] J. P. Haldar and J. Zhuo, “P-LORAKS: Low-rank modeling of local k-space neighborhoods with parallel imaging data,” Magn. Reson. Med., vol. 75, no. 4, pp. 1499-1514, 2016.
[10] J. C. Ye, Y. Han, and E. Cha, “Deep convolutional framelets: A general deep learning framework for inverse problems," SIAM J. Imaging Sci., vol. 11, no. 2, pp. 991-1048, 2018.
[11] T. Eo, H. Shin, Y. Jun, T. Kim, and D. Hwang, “Accelerating Cartesian MRI by domain-transform manifold learning in phase-encoding direction,” Med. Image Anal., vol. 63, 101689, 2020.
[12] A. Pramanik, H. Aggarwal, and M. Jacob, “Deep generalization of structured low-rank algorithms (Deep-SLR),” IEEE Trans. Med. Imaging, vol. 39, no. 12, pp. 4186-4197, 2020.
[13] K. Hammernik et al., “Learning a variational network for reconstruction of accelerated MRI data,” Magn. Reson. Med., vol. 79, no. 6, pp. 3055-3071, 2018.
[14] F. K. Zbontar et al., “FastMRI: An open dataset and benchmarks for accelerated MRI,” arXiv: 1811.08839, 2019.
[15] L. Ying and J. Sheng, “Joint image reconstruction and sensitivity estimation in SENSE (JSENSE),” Magn. Reson. Med., vol. 57, no. 6, pp. 1196-1202, 2007.
[16] Q. Yang, Z. Wang, K. Guo, C. Cai, and X. Qu, “Physics-driven synthetic data learning for biomedical magnetic resonance,” IEEE Signal Process. Mag., DOI: 10.1109/MSP.2022.3183809, 2022.
[17] Z. Wang et al., “A sparse model-inspired deep thresholding network for exponential signal reconstruction--Application in fast biological spectroscopy,” IEEE Trans. Neural Netw. Learn. Syst., DOI: 10.1109/TNNLS.2022.3144580, 2022.