
指数稀疏深度自适应阈值网络及快速磁共振波谱应用和智能云平台(中文,English)
王孜1, 郭迪2, 涂章仁1, 黄奕晖1, 周毅荣1, 王健1, 冯柳宾3, 林东海3, 游永富4, Tatiana Agback5, Vladislav Orekhov6, 屈小波1,*
1 厦门大学,电子科学系,福建等离子体与磁共振重点研究实验室,健康医疗大数据国家研究院,中国,厦门.
2 厦门理工学院,计算机与信息工程学院,中国,厦门.
3 厦门大学,化学与化工学院,中国,厦门.
4 中国移动,中国,厦门.
5 瑞典农业科学大学,分子科学系,瑞典,乌普萨拉.
6 哥德堡大学,化学与分子生物学系,瑞典,哥德堡.
* Emails: quxiaobo <at> xmu.edu.cn or quxiaobo2009 <at> gmail.com
引用
Zi Wang, Di Guo, Zhangren Tu, Yihui Huang, Yirong Zhou, Jian Wang, Liubin Feng, Donghai Lin, Yongfu You, Tatiana Agback, Vladislav Orekhov, Xiaobo Qu, A Sparse Model-inspired Deep Thresholding Network for Exponential Signal Reconstruction—Application in Fast Biological Spectroscopy, IEEE Transactions on Neural Networks and Learning Systems, doi: 10.1109/TNNLS.2022.3144580, 2022.
概要
指数信号的快速采集在雷达通信、荧光显微镜成像、电子系统的模数转换、地球科学、生物核磁共振波谱等领域被广泛使用。非均匀采样是一种常用的技术,它通过减少采集数据量来实现快速采集。由于数据采样不足,傅里叶变换得到的频谱中存在大量的伪影。因此,对欠采样指数信号的可靠重建是信号处理领域的前沿和重要问题之一。
深度学习在这一领域已经显示出惊人的潜力,但仍旧存在许多亟待解决的问题,如缺乏鲁棒性和可解释性,这极大地限制了其应用。因此,设计一种适合于指数信号重建的深度神经网络,实现更鲁棒的性能、更强的可解释性和更低的计算成本,是本工作的主要出发点。
主要内容
在本工作中,结合稀疏最优化和深度学习的优点,我们提出了一种深度自适应阈值网络,称为MoDern(图1)。它遵循求解稀疏模型的迭代重建过程来构建神经网络架构,并精心设计了一种可学习的软阈值模块来自适应地消除欠采样带来的频谱伪影。大量合成和生物实验数据的结果表明,MoDern突破了最先进方法的性能瓶颈,能够实现鲁棒、高保真和超快速的重建,同时网络的可解释性强。值得注意的是,MoDern的网络结构紧凑,网络参数少,并且仅使用指数函数的合成数据进行训练。由于其在训练和测试数据不匹配方面(如采样率和数据尺寸)展现的鲁棒性,它可以很好地在各种场景下对生物实验数据进行泛化,而无需重新训练。进一步地,我们将其扩展为一个开放获取、高性能和易于使用的云计算平台,称为XCloud-MoDern,以服务于更多的生物波谱研究人员,并为生物应用的发展提供了一个有前景的策略。

图1. MoDern和XCloud-MoDern的概述。(a)MoDern网络结构,(b)可学习自适应软阈值模块,(c)用于处理多维核磁共振生物波谱的人工智能云计算平台XCloud-MoDern的示意图。
在不同的指数信号个数(谱峰数)、采样率的合成数据的欠采样重建实验中(每种情况进行100次随机实验),相比与其他前沿方法,所提方法MoDern不仅保持了超快速重建的优势,还在指数个数较多或采样率较低的情况下,可以提供更低的整体重建误差和更小的能量损失(图2)。这表明了MoDern对一般性指数信号处理的重要意义。
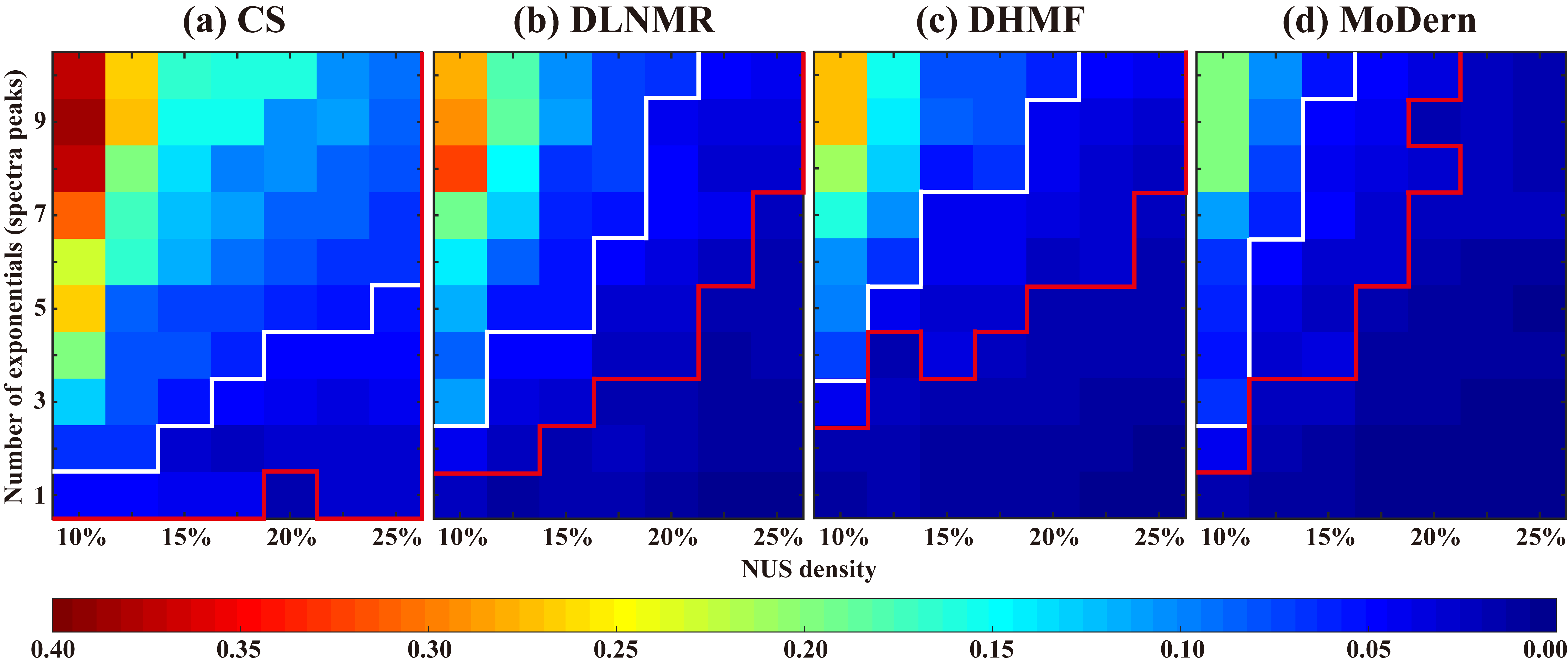
图2. 不同场景下合成数据的重建。(a)到(d)分别为CS、DLNMR、DHMF和MoDern的平均相对二范数重建误差(RLNE)。注:红色(或白色)线是设定的经验边界,代表误差阈值为0.02(或0.05)。在边界以下,区域的重建误差小于误差阈值。
核磁共振波谱在现代化学、生物和医学等领域具有极其重要的作用,但其数据采集时间长。快速采样和可靠重建是一种非常值得期待的技术。因此,我们进一步将所提方法MoDern推广到指数信号重建的重要应用——快速多维生物波谱中。
限制现有深度学习方法广泛应用于快速多维生物波谱的一个关键因素是缺乏鲁棒性。在实际应用中,它们很难克服训练数据和测试数据之间的不匹配,这意味着需要花费大量的时间对网络进行重新训练来处理不同场景下的各种任务。在这里,我们以采样率的不匹配问题为例,使用15%(或10%)采样率的数据集训练MoDern和DLNMR,来重建不同多种采样率下的二维(或三维)生物波谱数据。当采样率明显偏离训练中使用的情况时,MoDern依旧保持了高质量的重建性能,而前沿的深度学习方法DLNMR出现了明显的谱峰强度畸变、伪影和谱峰的相关系数降低(图3)。这表明了MoDern是一种鲁棒可靠的技术,可以在广泛的场景下有效工作。
更多MoDern在高动态范围、大蛋白、无序蛋白等挑战数据和代谢物相对浓度量化上的优异表现详见全文。
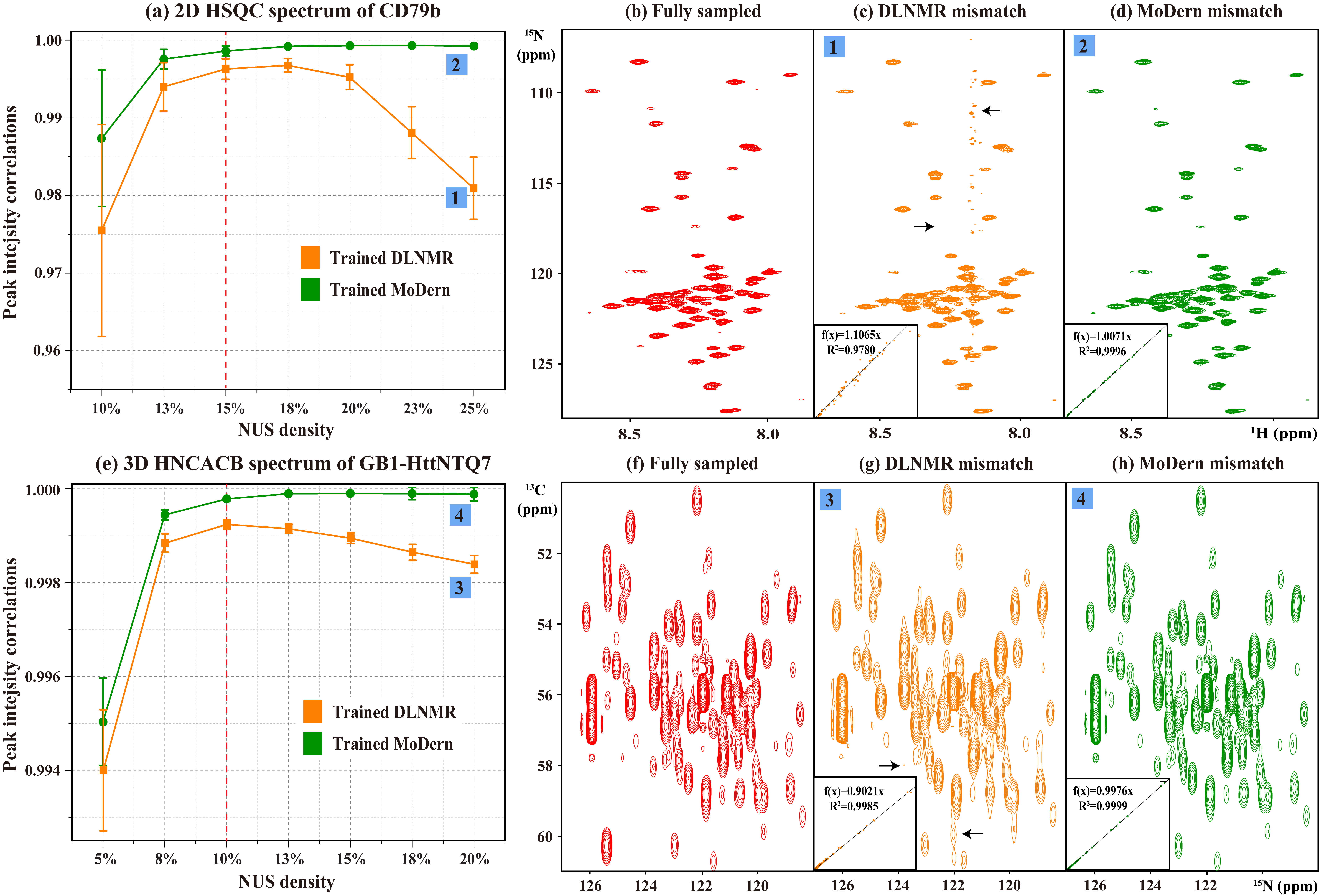
图3. 二维和三维生物波谱数据的不匹配重建。(a)CD79b蛋白的全采样的二维HSQC谱与重建谱之间的皮尔逊相关系数。(b)到(d)分别为全采样谱、DLNMR和MoDern的典型重建谱。(e)GB1-HttNTQ7蛋白的全采样的三维HNCACB谱与重建谱之间的皮尔逊相关系数。(f)到(h)分别为全采样谱、DLNMR和MoDern的典型重建谱的13C-15N平面投影的局部放大。注:(a)和(e)中的红色虚线表示网络训练时使用的采样率。
相关资料
致谢
本工作得到了国家自然科学基金(62122064、61971361、61871341、61811530021),福建省自然科学基金(2021J011184),国家重点研发计划(2017YFC0108703),厦门大学南强拔尖人才计划,瑞典研究委员会基金(2015–04614)和瑞典战略研究基金(ITM17-0218)的资助。
参考文献
[1] X. Qu, M. Mayzel, J.-F. Cai, Z. Chen, and V. Orekhov, “Accelerated NMR spectroscopy with low-rank reconstruction,” Angew. Chem. Int. Ed., vol. 54, no. 3, pp. 852-854, 2015.
[2] J. Ying, J.-F. Cai, D. Guo, G. Tang, Z. Chen, and X. Qu, “Vandermonde factorization of Hankel matrix for complex exponential signal recovery—Application in fast NMR spectroscopy,” IEEE Trans. Signal Process., vol. 66, no. 21, pp. 5520-5533, 2018.
[3] V. Jaravine, I. Ibraghimov, and V. Yu Orekhov, “Removal of a time barrier for high-resolution multidimensional NMR spectroscopy,” Nature Met., vol. 3, no. 8, pp. 605-607, 2006.
[4] M. Mobli and J. C. Hoch, “Nonuniform sampling and non-Fourier signal processing methods in multidimensional NMR,” Prog. Nucl. Magn. Reson. Spectrosc., vol. 83, pp. 21-41, 2014.
[5] Y. Huang, J. Zhao, Z. Wang, D. Guo, and X. Qu, “Exponential signal reconstruction with deep Hankel matrix factorization,” IEEE Trans. Neural Netw. Learn. Syst., 2021, DOI: 10.1109/TNNLS.2021.3134717.
[6] K. Kazimierczuk and V. Y. Orekhov, “Accelerated NMR spectroscopy by using compressed sensing,” Angew. Chem. Int. Ed., vol. 50, no. 24, pp. 5556-5559, 2011.
[7] T. Qiu, Z. Wang, H. Liu, D. Guo, and X. Qu, “Review and prospect: NMR spectroscopy denoising and reconstruction with Low-Rank Hankel matrices and tensors,” Magn. Reson. Chem., vol. 59, pp. 324-345, 2021.
[8] X. Qu et al., “Accelerated nuclear magnetic resonance spectroscopy with deep learning,” Angew. Chem. Int. Ed., vol. 59, no. 26, pp. 10297-10300, 2020.
[9] Y. LeCun, Y. Bengio, and G. Hinton, “Deep learning,” Nature, vol. 521, pp. 436-444, 2015.
[10] D. Chen, Z. Wang, D. Guo, V. Orekhov, and X. Qu, “Review and prospect: Deep learning in nuclear magnetic resonance spectroscopy,” Chem. Eur. J., vol. 26, no. 46, pp. 10391-10401, 2020.
[11] Z. Wang et al., “One-dimensional deep low-rank and sparse network for accelerated MRI," arXiv:2112.04721, 2021.
[12] J. Hoch and A. Stern, NMR Data Processing. 1996.
[13] D. L. Donoho, “De-noising by soft-thresholding,” IEEE Trans. Inf. Theory, vol. 41, no. 3, pp. 613-627, 1995.