屈小波1*, 黄奕晖1, 鲁恒发1, 邱天予1, 郭迪2, Vladislav Orekhov3, 陈忠1*
1厦门大学,电子科学系,福建等离子体与磁共振重点研究实验室,中国,厦门,361005
2厦门理工学院,计算机与信息工程学院,中国,厦门,361024
3哥德堡大学,化学与分子生物学学院,瑞典,哥德堡,40530
邮箱:屈小波(quxiaobo@xmu.edu.cn),陈忠(chenz@xmu.edu.cn)
引用:
Xiaobo Qu*, Yihui Huang, Hengfa Lu, Tianyu Qiu, Di Guo, Vladislav Orekhov,Zhong Chen*. Accelerated Nuclear Magnetic Resonance Spectroscopy with Deep Learning, Angewandte Chemie International Edition, DOI: 10.1002/anie.201908162,2019
概要:
核磁共振波谱是化学和生物学中不可缺少的工具,但其采样时间长。利用深度学习和神经网络,我们提出了一种快速可靠且高质量的核磁共振波谱重建方法。结果表明,神经网络的训练可以使用仿真核磁共振信号来实现,从而缓解了深度学习方法通常对大量真实数据的需求。
方法:
我们的方法DLNMR只使用仿真数据作为训练数据,这与许多使用真实数据作为训练数据的深度学习方法有很大不同。全采样频谱满足![]() ,其中
,其中![]() 是傅里叶变换,
是傅里叶变换,![]() 是全采样FID。欠采样FID服从
是全采样FID。欠采样FID服从![]() ,其中
,其中![]() 是欠采样算子。
是欠采样算子。
我们根据如下经典指数函数模型[1-5],对全采样FID x进行模拟:
 (1-1)
(1-1)
其共有J个指数构成,Aj, ![]() , τj 和fj 分别代表第jth个指数的幅度、相位、衰减常数和频率。
, τj 和fj 分别代表第jth个指数的幅度、相位、衰减常数和频率。
重建一个欠采样FID,等价于欠采样FID到目标谱的映射。如图1所示,在DLNMR中,神经网络被用于训练和学习这种映射。
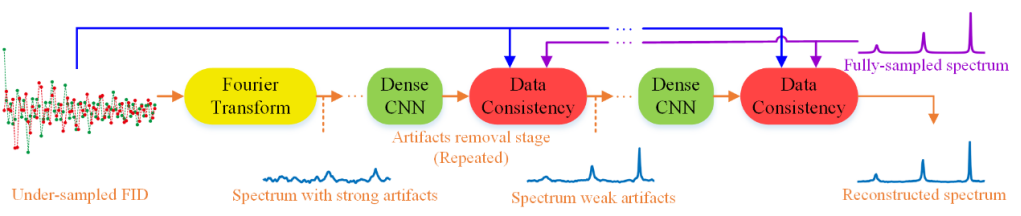
图1. 深度学习谱重建的流程图。
主要结果:
为了让DLNMR与经典的欠采样核磁共振波谱重建方法做比较,例如重建二维谱的低秩(Low Rank, LR)方法[2]和重建三维谱的压缩感知(Compressed Sensing, CS)方法[6, 7],一些二维和三维蛋白谱被用于重建和评估。结果显示,DLNMR、LR和CS方法均获得非常高的峰值强度相关系数。其中二维谱的采样率为25%,三维谱的采样率为10%。图2到图6显示了重建的高保真度。更多不同的重建谱详见发表的文章。
1. 二维谱重建:

图2. 重建B细胞受体CD79b蛋白的二维1H–15N HSQC谱。(a)至(c)分别是全采样波谱、采样率25%的LR和DLNMR重建波谱;(d)和(e)分别是LR和DLNMR重建波谱的峰值强度相关系数;(f)和(e)是放大对的一维15N谱,红色、黄色和绿色线分别代表全采样和LR、DLNMR重建的波谱。
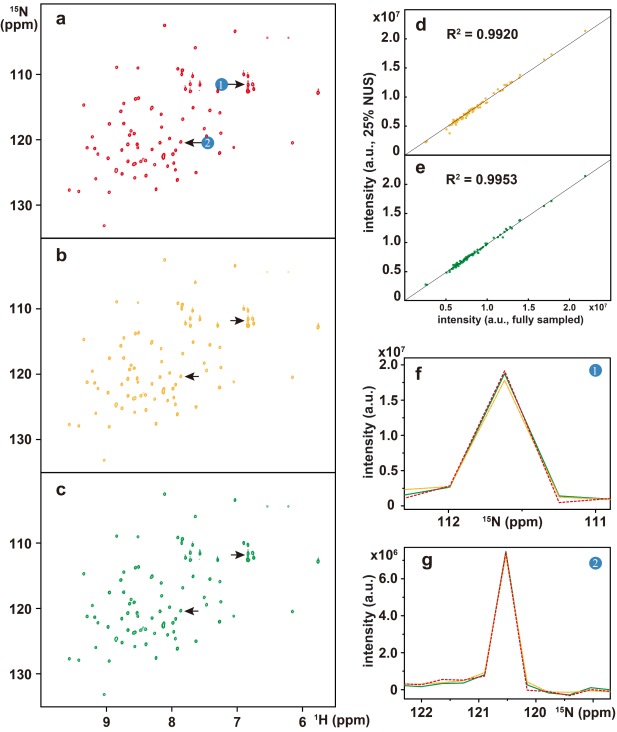
图3. 重建泛素的二维1H–15N HSQC谱。(a)至(c)分别是全采样波谱、采样率25%的LR和DLNMR重建波谱;(d)和(e)分别是LR和DLNMR重建波谱的峰值强度相关系数;(f)和(e)是放大对的一维15N谱,红色、黄色和绿色线分别代表全采样和LR、DLNMR重建的波谱。
2. 三维谱重建
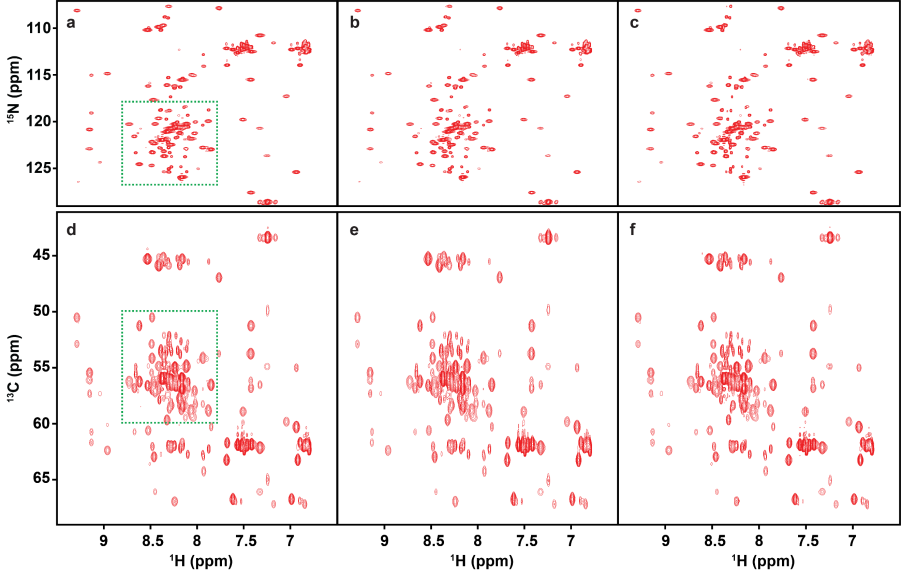
图4. 三维HNCACB的1H-15N和1H-13C平面投影谱。(a)(d)为全采样谱,(b)(e)和(c)(f)分别为采样率10%的CS和DLNMR重建谱。注:绿色虚线所指的矩形投影区域如图5所示。
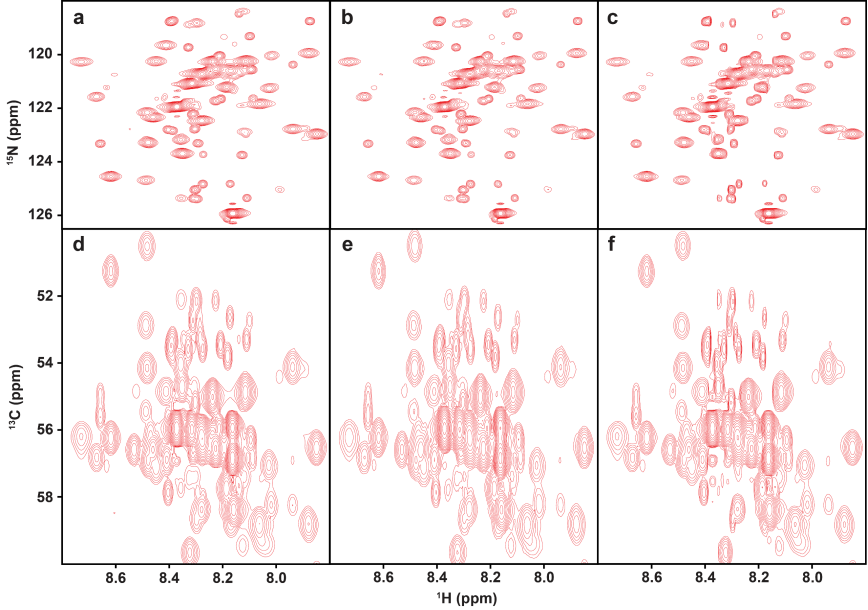
图 5. 三维HNCACB的1H-15N和1H-13C平面投影谱的子区域。(a)(d)为全采样谱,(b)(e)和(c)(f)分别为采样率10%的CS和DLNMR重建谱。
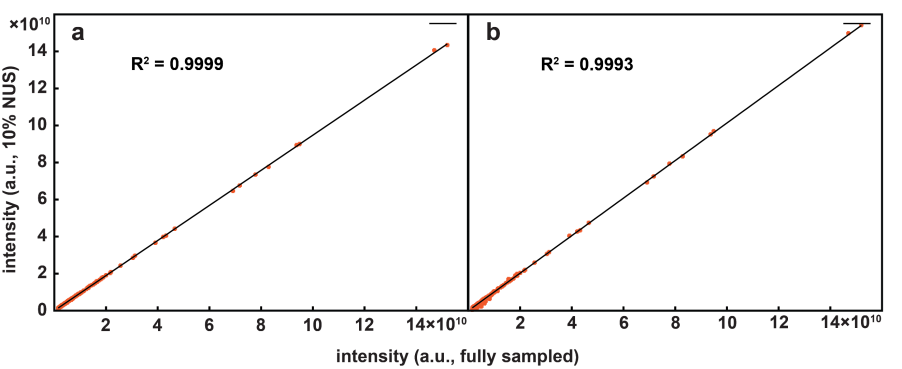
图 6. 全采样3D HNCACB和图5所示的重建波谱的相关系数。(a)和(b)分别为CS和DLNMR重建波谱的峰值强度相关系数。
3. 低采样率重建
在较低的采样率(10%和15%)下,DLNMR在100次随机采样试验中展示了较高的相关性和较低的相关系数波动程度。图7 (a)和(b)展示了在低采样率下,DLNMR能得到更高质量的波谱。这些观察结果表明,与LR方法相比,DLNMR可以显著节省采样时间,并且在不同的采样率下更为稳健,进而获得更稳定的重建。

图 7. 在不同采样率下二维1H-15N HSQC谱的重建结果。(a)至(d)分别为10%、15%、20%和25%采样率下的重建结果。绿色和黄色标记分别代表DLNMR和LR方法。
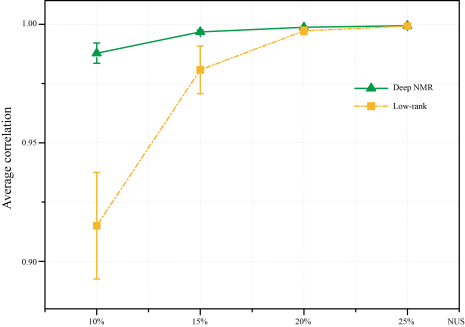
图 8. 在不同采样率下二维1H-15N HSQC谱的相关系数。注:绿色线和黄色线分别代表DLNMR和LR方法与全采样波谱进行比较的皮尔逊相关系数R2。误差条是100次随机重采样试验中相关系数的标准差。

图 9. 在不同采样率下其他二维谱的相关系数。注:绿色线和黄色线分别代表DLNMR和LR方法与全采样波谱进行比较的皮尔逊相关系数R2。误差条是100次随机重采样试验中相关系数的标准差。
4. 重建时间
在不影响重建波谱质量的情况下,DLNMR比其他优秀的传统方法(如LR[2]和CS[8])要快得多。比较结果表明,DLNMR的重建速度比传统算法快几个数量级。
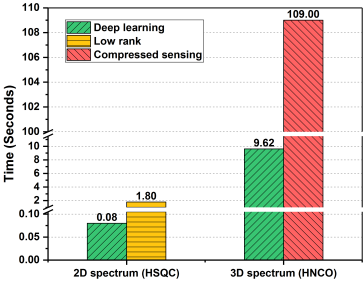
图 10. DLNMR、LR和CS的重建时间。实验在一台双12核CPU(2.2GHz)计算机服务器上进行,该服务器配有128GB RAM和Nvidia Tesla K40M,分别在Tensorflow(GPU)、MATLAB(CPU)和MddNMR(CPU)上实现了DLNMR、LR和CS。基于CPU的24线程并行计算加速了LR和CS算法。被重建二维谱的间接维数为256点,直接维数为116点。三维谱的间接维数为60x60点,直接维数为732点。
下载:
文章:
CSG网站: 这里
预印本: https://arxiv.org/abs/1904.05168
官方: https://doi.org/10.1002/anie.201908162
数据:
1) 二维谱
数据
GB1的HSQC谱,原始数据的下载地址为https://www.ibbr.umd.edu/nmrpipe/demo.html
2) 三维谱
数据
天青蛋白的HNCO谱[9],原始数据的下载地址为http://mddnmr.spektrino.com/download
致谢:
1) 数据提供者
作者感谢Marius Clore和Samuel Kotler提供了三维 HNCAB波谱;Jinfa Ying协助处理三维 HNCAB波谱并进行了有益的讨论;Luke Arbogast和Frank Delaglio提供了GB1的二维HSQC波谱。
2) 基金
这项工作得到了国家自然科学基金(61571380, 61871341和U1632274),瑞典国家自然科学基金国际合作高等教育基金61811530021,中国国家重点研究开发计划2017YFC00108703,福建省自然科学基金2018J06018、中央大学基础研究基金20720180056、厦门市科技计划3502Z20183053、国家留学基金委201806315010、201808350010,以及瑞典研究委员会2015-04614的资助。
参考文献:
[1] J. C. Hoch and A. Stern, NMR Data Processing. Wiley, 1996.
[2] X. Qu, M. Mayzel, J.-F. Cai, Z. Chen, and V. Orekhov, "Accelerated NMR Spectroscopy with Low-Rank Reconstruction," Angewandte Chemie, International Edition, vol. 54, no. 3, pp. 852-854, 2015.
[3] H. M. Nguyen, X. Peng, M. N. Do, and Z. Liang, "Denoising MR Spectroscopic Imaging Data with Low-Rank Approximations," IEEE Transactions on Biomedical Engineering, vol. 60, no. 1, pp. 78-89, 2013.
[4] J. Ying, H. Lu, Q. Wei, J. Cai, D. Guo, J. Wu, Z. Chen, and X. Qu, "Hankel Matrix Nuclear Norm Regularized Tensor Completion for N-dimensional Exponential Signals," IEEE Transactions on Signal Processing, vol. 65, no. 14, pp. 3702-3717, 2017.
[5] J. Ying, J. Cai, D. Guo, G. Tang, Z. Chen, and X. Qu, "Vandermonde Factorization of Hankel Matrix for Complex Exponential Signal Recovery—Application in Fast NMR Spectroscopy," IEEE Transactions on Signal Processing, vol. 66, no. 21, pp. 5520-5533, 2018.
[6] Y. Pustovalova, M. Mayzel, and V. Y. Orekhov, "XLSY: Extra-Large NMR Spectroscopy," Angewandte Chemie, International Edition, vol. 57, no. 43, pp. 14043-14045, 2018.
[7] X. Qu, X. Cao, D. Guo, and Z. Chen, " Compressed Sensing for Sparse Magnetic Resonance Spectroscopy," International Society for Magnetic Resonance in Medicine 18th Scientific Meeting (ISMRM), Stockholm, Sweden, May 1-7, pp. 3371, 2010.
[8] K. Kazimierczuk and V. Y. Orekhov, "Accelerated NMR Spectroscopy by Using Compressed Sensing," Angewandte Chemie, International Edition, vol. 50, no. 24, pp. 5556-5559, 2011.
[9] D. M. Korzhnev, B. G. Karlsson, V. Y. Orekhov, and M. Billeter, "NMR Detection of Multiple Transitions to Low-Populated States in Azurin," Protein Science, vol. 12, no. 1, pp. 56-65, 2003.