基于低秩和k空间数据一致性的多通道磁共振图像重建方法 (中文,English)
张心林1, 郭迪2, 黄以满1, 陈颖1, 王连生3, 黄峰4, 徐勤4, 屈小波1,*
1 厦门大学,电子科学系,福建等离子体与磁共振重点研究实验室,中国,厦门,361005.
2 厦门理工学院,计算机与信息工程学院,中国,厦门,361024.
3 厦门大学,信息科学与工程学院,361005.
4 东软医疗系统有限公司,中国,上海,200241.
* Emails: quxiaobo <at> xmu.edu.cn or quxiaobo2009 <at> gmail.com
引用
Xinlin Zhang, Di Guo, Yiman Huang, Ying Chen, Liansheng Wang, Feng Huang, Qin Xu, Xiaobo Qu, Image Reconstruction with Low-rankness and Self-consistency of k-space Data in Parallel MRI, Medical Image Analysis, 63: 101687, 2020.
摘要
并行成像已经成为一种有效并且被广泛应用的加速磁共振成像(Magnetic resonance imaging, MRI)数据采集的技术。稀疏采样的出现提供了更大倍数的加速,同时采样方式更加灵活,重建图像误差更小。然而,从有限的数据重建MRI图像仍然是一项艰巨的任务。近年来利结构化低秩矩阵重建MRI图像的方法,在提供高质量图像方面具有优势。然而,它们都没有采用常规获取的校准数据来改善并行磁共振成像的重建图像质量。
本文提出了一种基于低秩和k空间数据一致性的并行成像MRI重建方法(STDLR-SPIRiT),利用k空间数据双方向的结构化低秩特性,以及多通道k空间数据间的一致性来重建图像。实验结果表明,在抑制伪影和降低误差方面,所提方法优于目前MRI重建最新方法。此外,当并行成像中自动校准信号受到限制时,所提出的方法仍能可靠重建图像。
方法
1. 背景
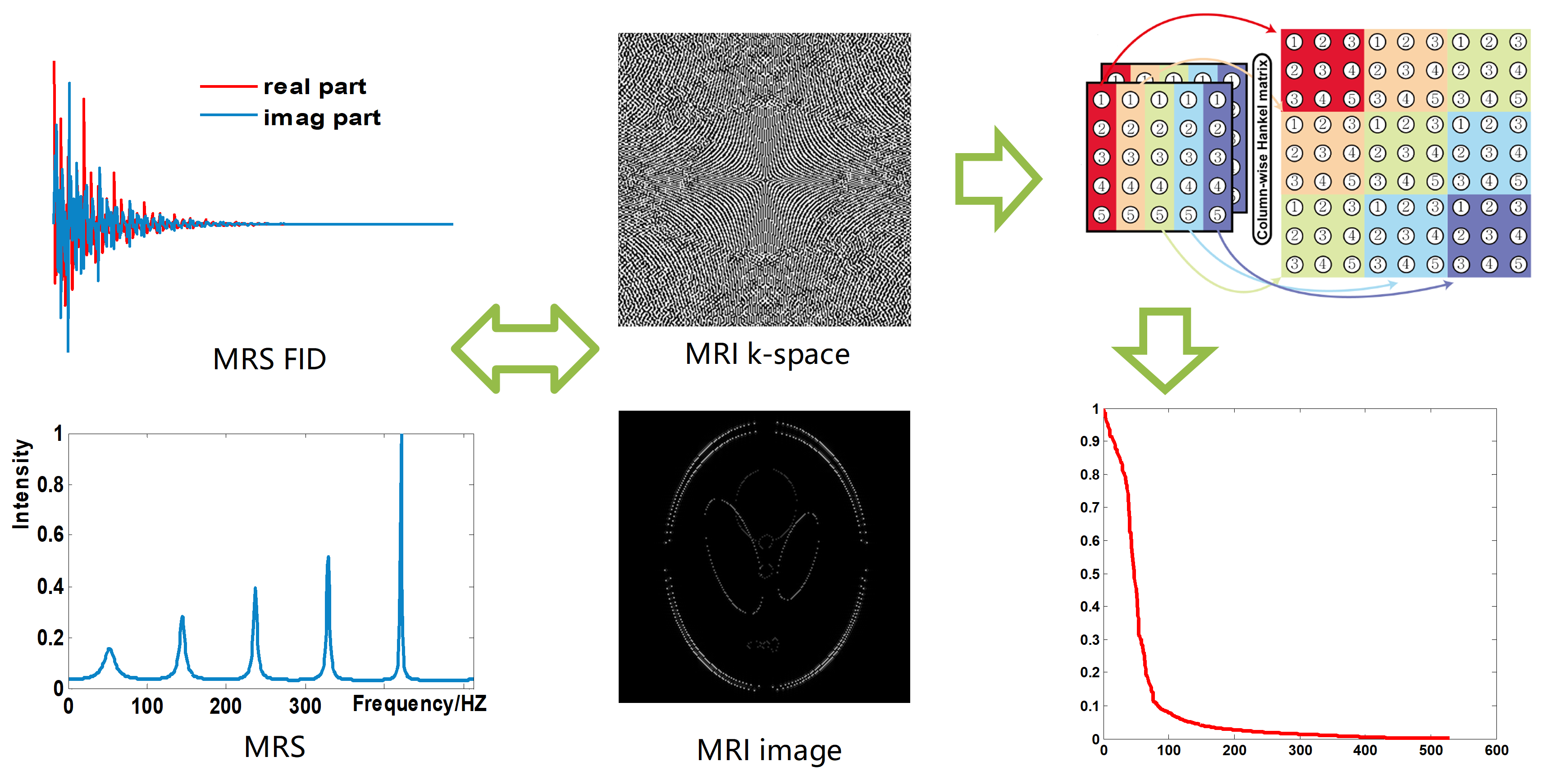
过长的采集时间给MRI的应用带来了挑战。并行成像技术显著缩短了扫描时间,但是这类方法需要采集足够的自动校准信号(Auto-calibration signal, ACS)。稀疏采样及重建是另一种加快数据采集的有效方法。重建方法可以分为两大类:稀疏方法和低秩方法。低秩结构化矩阵方法是一类最新的低秩重建方法,最开始被应用在磁共振波谱(Magnetic resonance spectroscopy, MRS)重建中。这类方法将MRS时域自由感应衰减信号(Free induction decay, FID)建模为指数函数的叠加,通过约束由FID构成的Hankel矩阵低秩性进行重建,显示出卓越的重建结果。近年来,MRS的指数函数性质逐步推广到MRI中,即将MRI中k空间建模为指数函数的叠加,约束k空间构成的结构化矩阵进行重建(图1)。低秩结构化矩阵的MRI重建具有误差低、对于采样模板鲁棒等优势。但是,这类方法仍然存在局限性,例如,ALOHA中分别约束水平和垂直方向的低秩导致重建结果不理想,以及现有的低秩结构化矩阵方法没有利用常见的ACS来提升重建结果。本文提出了一种k空间域重建方法,该方法同时利用了k空间结构化低秩特性和ACS数据一致性。

图 1. MRS和MRI中的结构化低秩性。
2. 基于低秩和k空间数据一致性的多通道磁共振图像重建方法(STDLR-SPIRiT)
已经证明,图像的稀疏性与k空间数据的低秩密切相关(图2)。通过约束k空间加权结构化矩阵的低秩性,可以实现k空间丢失数据的恢复。我们提出了一种重建方法,利用k空间数据双方向低秩特性,以及ACS数据一致性来重建图像:
$$ \left( \mathbf{STDLR-SPIRiT}\right)\quad \underset{\mathbf{X}}{\mathop{\min }}\,\sum\limits_{i}{{{\left\| \tilde{\mathcal{H}}{{\mathcal{W}}_{i}}\mathbf{X} \right\|}_{*}}}+\frac{{{\lambda }_{1}}}{2}\left\| \mathcal{G}\mathbf{X}-\mathbf{X} \right\|_{F}^{2}+\frac{{{\lambda }_{2}}}{2}\left\| \mathbf{Y}-\mathcal{U}\mathbf{X} \right\|_{F}^{2}, $$ 这里$i$表示$=$或者$\bot $,即水平或垂直方向。$\mathbf{X}=\left[ {{\mathbf{X}}_{1}},{{\mathbf{X}}_{2}},\cdots ,{{\mathbf{X}}_{J}} \right]$表示待重建的多通道k空间数据,$\mathbf{Y}=\left[ {{\mathbf{Y}}_{1}},{{\mathbf{Y}}_{2}},\cdots ,{{\mathbf{Y}}_{J}} \right]$表示对采集信号进行非采样点位置填零的数据,$\mathcal{U}$表示欠采样并在未采样点进行填零操作的算子。$\tilde{\mathcal{H}}{{\mathcal{W}}_{i}}\mathbf{X}=\left[ \mathcal{H}{{\mathbf{W}}_{i}}\odot {{\mathbf{X}}_{1}},\cdots ,\mathcal{H}{{\mathbf{W}}_{i}}\odot {{\mathbf{X}}_{J}} \right]$,这里$\mathcal{H}$表示将矩阵转化为结构化矩阵的算子,${{\mathbf{W}}_{i}}$表示权重矩阵,其权重为滤波器在水平或垂直方向上的傅里叶变换。$\mathcal{G}$表示校准数据一致性的卷积核算子。${{\left\| \cdot \right\|}_{*}}$表示对加权结构化矩阵施加低秩约束,${{\lambda }_{1}}$和${{\lambda }_{2}}$是权衡低秩约束、ACS数据一致性以及采样数据一致性的参数。STDLR SPIRiT的示意图如图3所示。
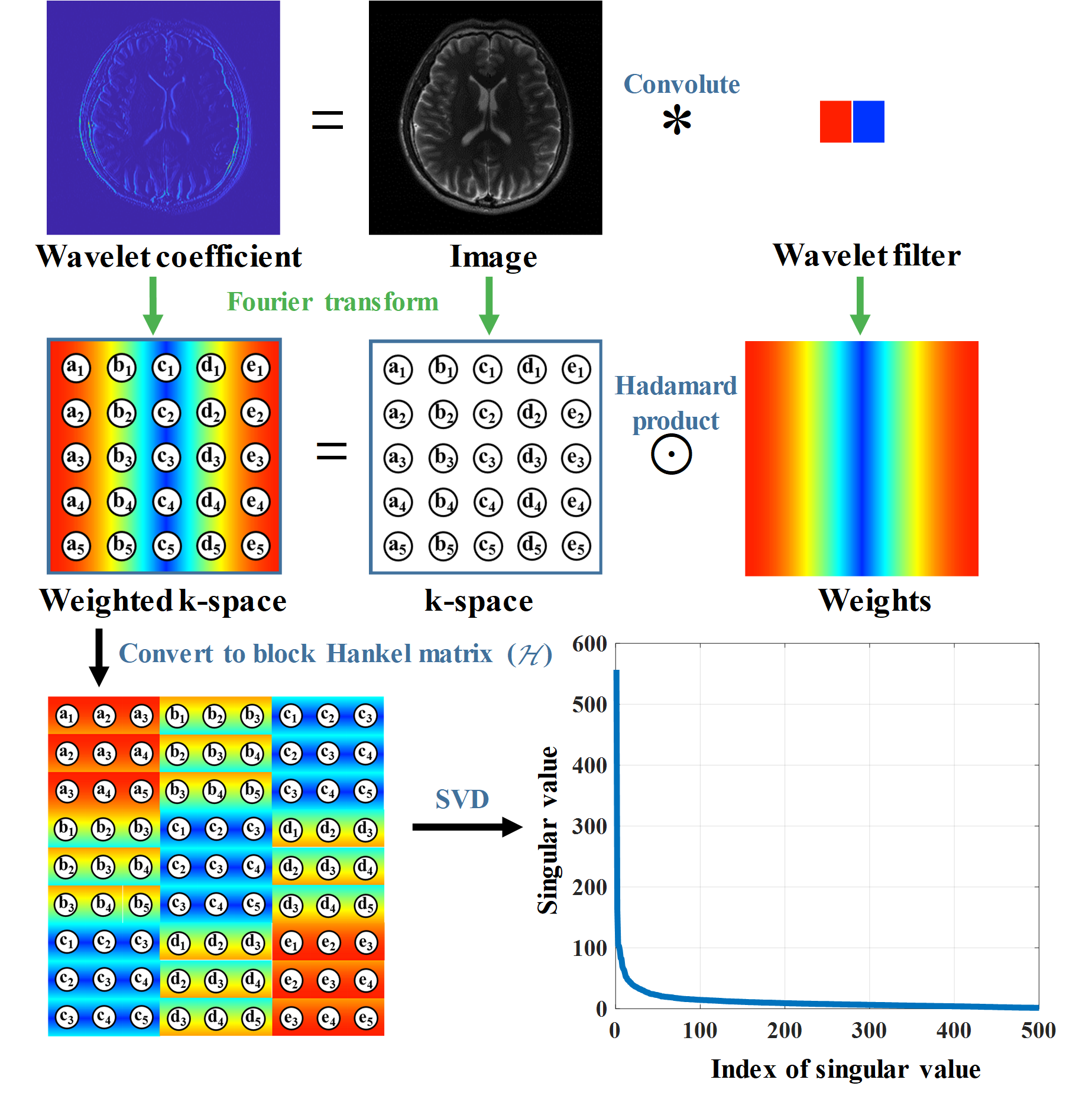
图 2. 加权k空间的低秩特性示意图。
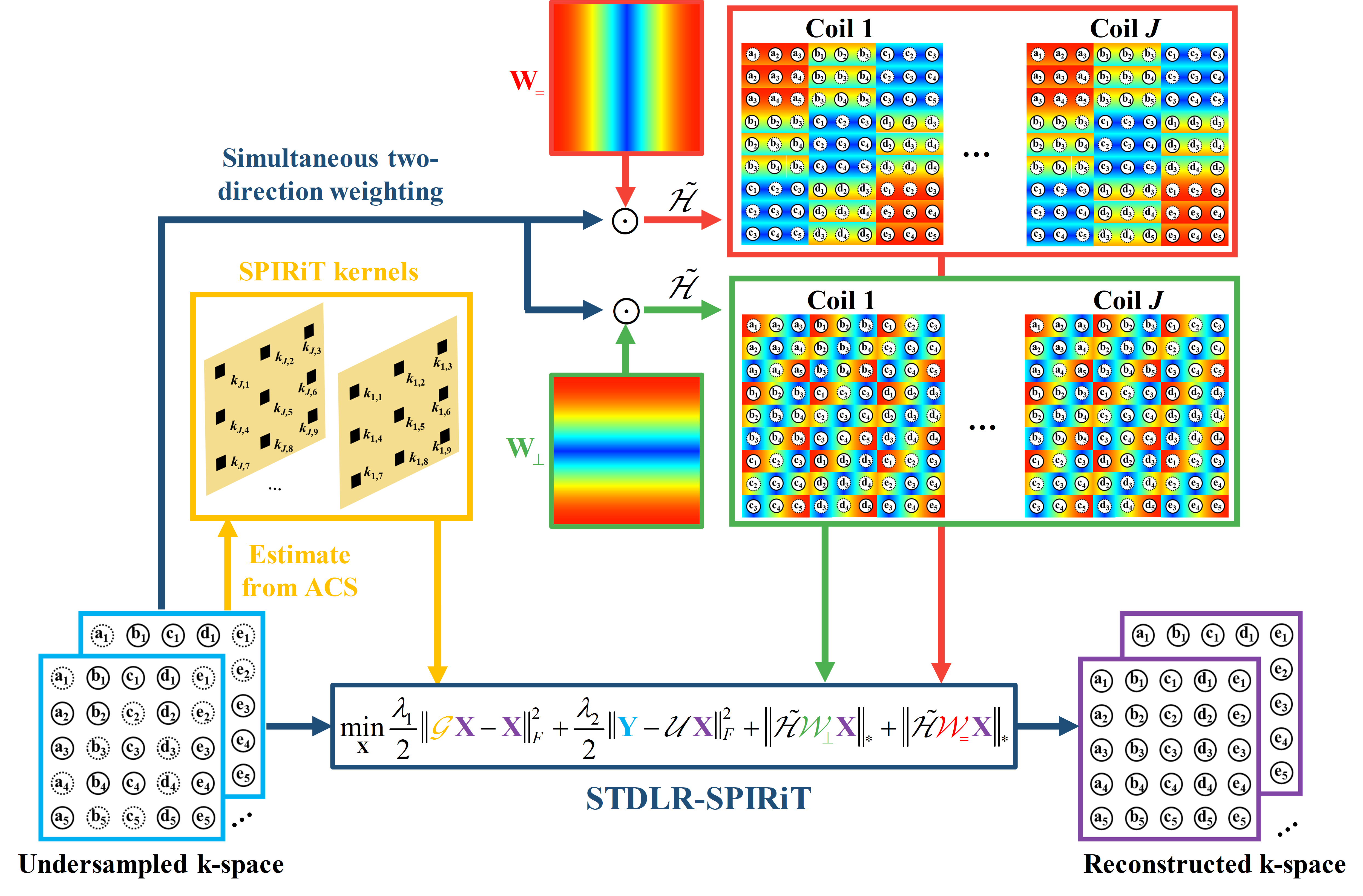
图 3. 所提方法的流程图。
3. 主要结果
我们将所提方法与GRAPPA、$\ell_1$-SPIRiT和ALOHA三种最新的重建方法进行了对比。实验数据为一组4通道实测人脑MRI图像,我们将对比3倍加速下的重建结果,这里我们将采用相对二范数误差(Relative L2-norm error, RLNE)作为指标来评价重建的结果,RLNE越低,则表示重建图像与原图之间的误差越小。
如图4所示,GRAPPA(图4 (b))和ALOHA(图4 (d))的重建图像有明显的伪影,而$\ell_1$-SPIRiT(图4 (c))和STDLR SPIRiT(图4 (e))的重建结果具有更好的伪影抑制效果。但是,在局部放大切片中,我们可以发现$\ell_1$-SPIRiT重建图像中存在较强的噪声和模糊。所提方法STDLR-SPIRiT的重建图像具有更好的信噪比和细节保持。此外,如图5所示,STDLR SPIRiT对ACS数据的数量显示出鲁棒性。
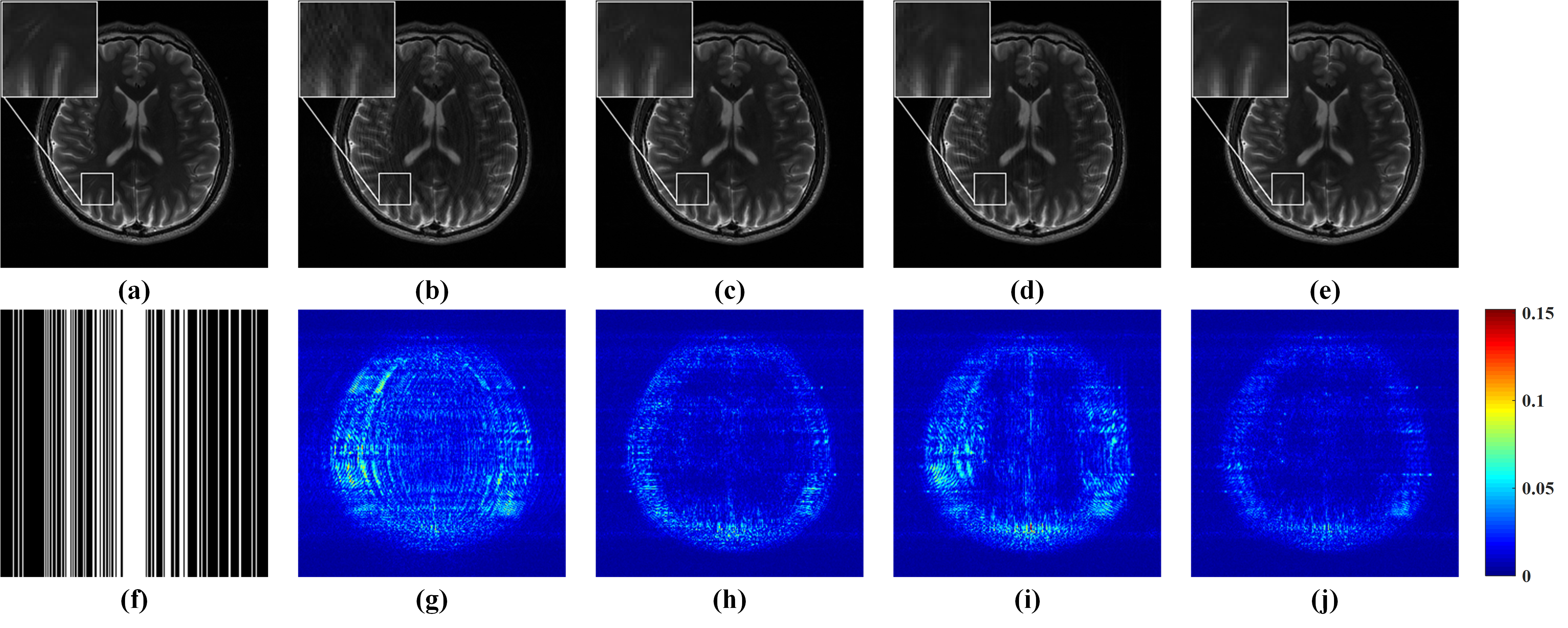
图 4. 4通道3倍加速人脑数据重建结果及误差。(a)全采样数据的SSOS图像;(b-e)分别由GRAPPA、$\ell_1$-SPIRiT、ALOHA和STDLR-SPIRiT重建结果的SSOS图像;(f)采样率为0.34的高斯-笛卡尔欠采样模板;(g-j)相应的重建误差分布图。(b-e)图的RLNE分别为:0.1335,0.0866,0.1117,0.0735.
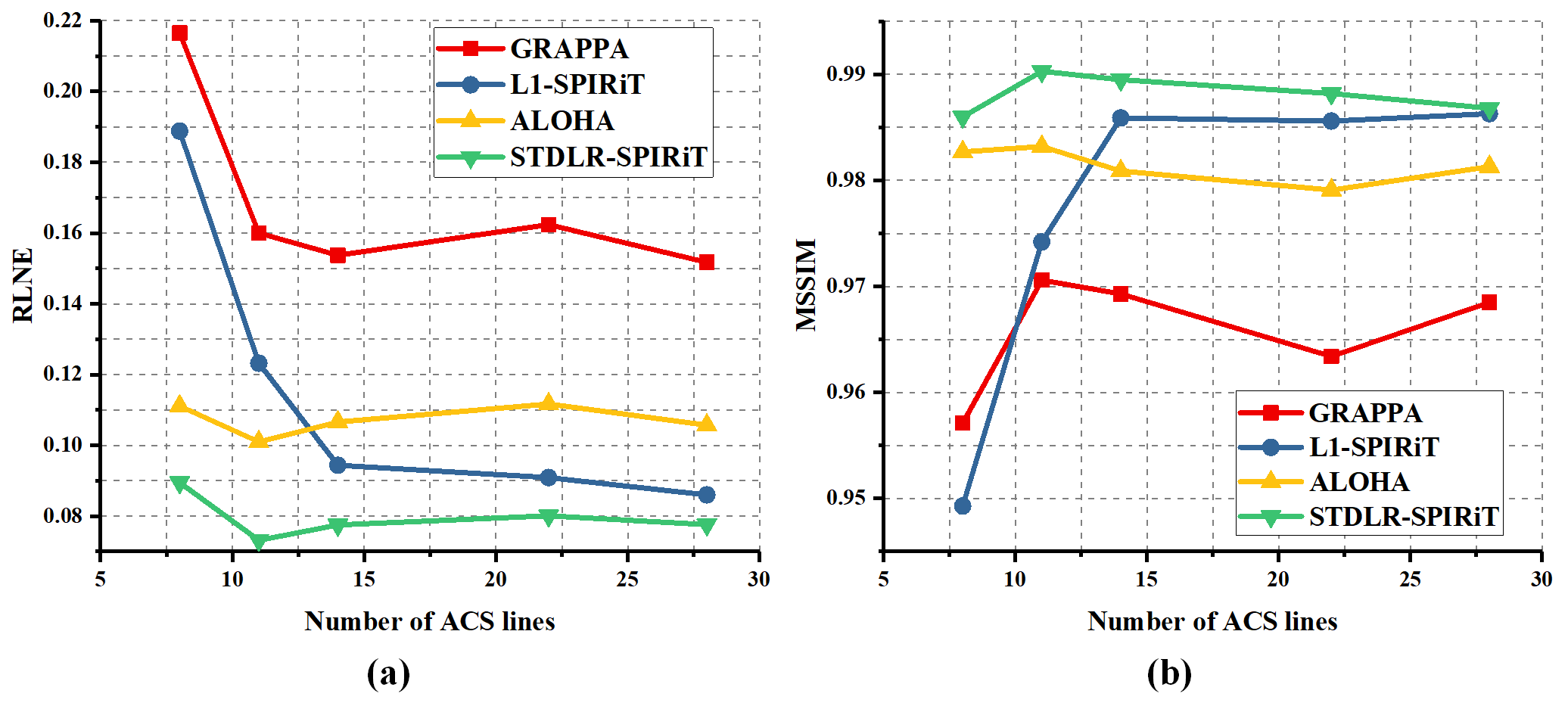
图 5. RLNE(a)和MSSIM(b)随ACS数量的变化趋势。这项实验是在一系列高斯-笛卡尔欠采样模板下进行的,采样率均为0.34,每个采样模板之间的差异是ACS的数量。
代码下载
所提方法的MATLAB代码可以从这里下载。
致谢
这项工作得到了国家重点研发计划(2017YFC0108700),国家自然科学基金(61971361、61871341、61811530021 U1632274,、61672335和61671399),福建省自然科学基金(2018J06018),中央高校基本科研基金(20720180056),厦门市科学技术计划(3502Z20183053)、国家留学基金和厦门大学南强拔尖人才计划的资助。
参考文献:
[1] J. Hamilton et al., “Recent advances in parallel imaging for MRI,” Progress in Nuclear Magnetic Resonance Spectroscopy, vol. 101, pp. 71–95, 2017.
[2] K. P. Pruessmann et al., “SENSE: Sensitivity encoding for fast MRI,” Magnetic Resonance in Medicine, vol. 42, pp. 952–962, 1999.
[3] M. A. Griswold et al., “Generalized autocalibrating partially parallel acquisitions (GRAPPA),” Magnetic Resonance in Medicine, vol. 47, no. 6, pp. 1202–1210, 2002.
[4] M. Lustig and J. M. Pauly, "SPIRiT: Iterative self-consistent parallel imaging reconstruction from arbitrary k-space," Magnetic Resonance in Medicine, vol. 64, no. 2, pp. 457-71, 2010.
[5] P.J.Shin et al., “Calibrationless parallel imaging reconstruction based on structured low-rank matrix completion,” Magnetic Resonance in Medicine, vol. 72, no. 4, pp. 959–970, 2014.
[6] K. H. Jin et a.l, “A general framework for compressed sensing and parallel mri using annihilating filter based low-rank hankel matrix,” IEEE Transactions on Computational Imaging, vol. 2, no. 4, pp. 480–495, 2016.
[7] J. P. Haldar, "Low-rank modeling of local k-space neighborhoods (LORAKS) for constrained MRI," IEEE Transaction on Medical Imaging, vol. 33, no. 3, pp. 668-81, 2014.
[8] G. Ongie and M. Jacob, "Off-the-grid recovery of piecewise constant images from few Fourier samples," SIAM Journal on Imaging Sciences, vol. 9, no. 3, pp. 1004-1041, 2016.
[9] X. Qu et al., "Accelerated NMR spectroscopy with low-rank reconstruction," Angewandte Chemie International Edition, vol. 54, no. 3, pp. 852-854, 2015.
[10] J. Ying, et al., "Hankel matrix nuclear norm regularized tensor completion for N-dimensional exponential signals," IEEE Transactions on Signal Processing, vol. 65, no. 14, pp. 3702-3717, 2017.